一種肉類檢測試劑盒和方法與流程
2023-06-26 23:06:36 2
本發明涉及一種肉的種類的檢測技術,特別是肉類檢測試劑盒和方法。
背景技術:
犛牛主要分布在中國西南部的高原及周邊的高山及亞高山地區,該地區海拔2000-5000m,環境艱苦惡劣。中國境內分布的犛牛佔世界犛牛總數的94%,但只佔了中國1.4億頭牛的很少部分。犛牛能利用這些地區的草場資源,提供肉、奶、毛和奶酪給該地區的人群。由於其獨特性,犛牛肉產品的價格遠高於當地的其他肉類價格,例如犛牛肉產品價格大概為黃牛肉價格的2倍。因此,近年來假犛牛肉製品持續增長,已佔到市場犛牛肉製品價格的80%。此外,有研究報告指出,西方國家市場上的犛牛肉製品價格也大幅增長,市場上錯誤標籤的產品也佔到15-39%。
由於肉類食品市場上的肉類真實性認證的需求,近年來科研工作者開發了許多肉類鑑定的方法。多數運用PCR-RFLP進行鑑定。但這類方法能鑑定的肉類品種有限,根據陳的研究小組的結果,他們的方法可以鑑定5個動物品種,並且鑑定混合樣品時只能鑑定3個品種。此外,Naue等在最近還發展了高解析度熔解曲線分析(HRM)方法,該方法能鑑定包括犛牛在內的28個動物品種,但其使用卻受到實驗程序的限制。例如該方法受到基因組DNA的總量、嚴格的緩衝液條件以及當給定基因存在未知的SNP位點的時候可能會在適時螢光定量PCR導致HRM值在改變,並隨之出現額外的檢測錯誤。
技術實現要素:
為了解決上述問題,本發明提供了一種新的肉類檢測試劑盒和方法。
本發明肉類檢測試劑盒,包含SEQ ID NO:1~2所示的引物對1、SEQ ID NO:3~4所示的引物對2、SEQ ID NO:5~6所示的引物對3、SEQ ID NO:7~8所示的引物對4、SEQ ID NO:9~10所示的引物對5、SEQ ID NO:11~12所示的引物對6、SEQ ID NO:13~14所示的引物對7、SEQ ID NO:15~16所示的引物對8和/或SEQ ID NO:17~18所示的引物對9。
本發明還提供了SEQ ID NO:1~2所示的引物對1、SEQ ID NO:3~4所示的引物對2、SEQ ID NO:5~6所示的引物對3、SEQ ID NO:7~8所示的引物對4、SEQ ID NO:9~10所示的引物對5、SEQ ID NO:11~12所示的引物對6、SEQ ID NO:13~14所示的引物對7、SEQ ID NO:15~16所示的引物對8和/或SEQ ID NO:17~18所示的引物對9在鑑別肉類中的用途。
其中,所述肉類是黃牛肉、犛牛肉、水牛肉、山羊肉、狗肉、豬肉、兔子肉、雞肉和鴨肉中的任意一種或者任意多種。
本發明還提供了一種肉類的檢測方法,包括如下步驟:
a,提取樣本DNA:提取待檢樣本中的DNA;
b,基因擴增:用權利要求1所述的試劑盒對待檢樣本中的DNA進行擴增;
c,結果檢測:對DNA擴增結果進行檢測。
其中,步驟a所述的樣本為黃牛肉、犛牛肉、水牛肉、山羊肉、狗肉、豬肉、兔子肉、雞肉和鴨肉中的任意一種或者任意多種。
本發明還提供了SEQ ID NO:1~2所示的引物對1、SEQ ID NO:3~4所示的引物對2、SEQ ID NO:5~6所示的引物對3、SEQ ID NO:7~8所示的引物對4、SEQ ID NO:9~10所示的引物對5、SEQ ID NO:11~12所示的引物對6、SEQ ID NO:13~14所示的引物對7、SEQ ID NO:15~16所示的引物對8和/或SEQ ID NO:17~18所示的引物對9在製備鑑別肉類的試劑中的用途。
其中,所述試劑是鑑別黃牛肉、犛牛肉、水牛肉、山羊肉、狗肉、豬肉、兔子肉、雞肉和/或鴨肉中的試劑。
本發明建立了一種鑑定包括通常在青藏高原地區才能發現的犛牛在內的動物品種的鑑定方法。該方法是肉類市場所急需要的一種檢測方法,可以檢測mt DNA含量不同的混合樣品。該方法需要具有精確性和和重複性以確保該方法在市場肉類產品鑑定中的權威性,並且該方法應該能夠在不同的品種和地理區域進行驗證。
本發明引物、試劑盒以及多重PCR檢測方法,能夠鑑定包括青藏高原地區在內的中國西南部動物肉類的方法,應用前景良好。
以下通過實施例形式的具體實施方式,對本發明的上述內容作進一步的詳細說明。但不應將此理解為本發明上述主題的範圍僅限於以下的實施例。凡基於本發明權利要求書記載的內容所實現的技術均屬於本發明的範圍。
附圖說明
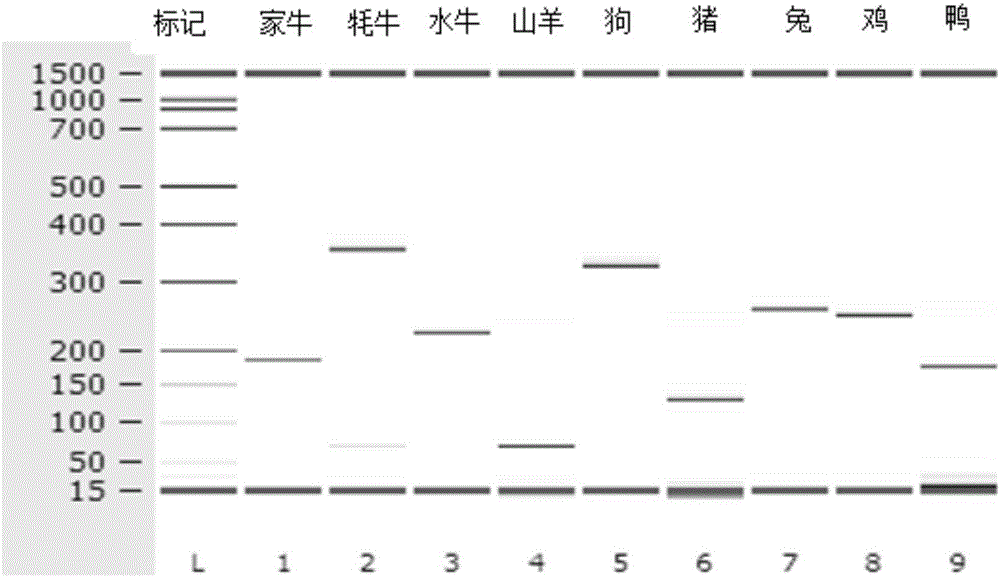
圖1多重PCR在Agilent 2100 DNA晶片上的鑑定結果,標記:Agilent 2100 DNA marker;
圖2犛牛和家牛線粒體DNA混合物在多重PCR中的檢驗結果;
圖3特異性檢測結果,其中,Ca:家牛,Ya:犛牛,Bu:水牛,Go:山羊,Do:狗,Pig:豬,Ra:兔,Ch:雞,Du:鴨。
具體實施方式
本發明中,除非特別說明,所有的操作都按生產商的推薦步驟進行。
實施例1本發明檢測方法
一、檢測方法
1、樣品準備,基因組DNA提取
本實驗從中國西南部的9種動物共採集到72份肉類樣品,包括:黃牛(川西北藏區,n=8)、犛牛(川西北藏區,n=8),s水牛(四川攀西地區,n=8)、藏山羊(川西北藏區,n=8)、狗(貴州銅仁,n=8)、豬(成都平原,n=8)、兔(成都平原,n=8)、雞(成都平原,n=8)、鴨(成都平原,n=8)見附表1。基因組DNA用Qiagen公司的Qiagen Mini Kit(Qiagen,Hilden,Germany)進行提取,然後溶於水中。由於在基因組DNA中線粒體DNA含量較少,我們用Qiagen公司的REPLI-g線粒體DNA試劑盒從提取的基因組DNA中擴增線粒體DNA。
一般而言,典型的實驗結果是50ul的反應體系中產生3~4ug/ul的線粒體DNA,肉乾產品用蒸餾水漂洗過夜然後用Qiagen Mini Kit.進行提取。
2、序列特異引物設計
通過分析12S rRNA基因序列,設計的引物序列及相關信息如下表1所示:
我們通過單獨的PCR和針對其他物種的交叉反應測試對引物的功能進行了判定。PCR反應在20μL的反應體系中進行,成分如下:1μL DNA模板、10mmol/L Tris-HCl(pH 8.3)緩衝液、2.5mmol/L MgCl2、50mmol/L KCl、10pmol/L引物、和1單位的TaqDNA聚合酶(TsingKe biotech co.,Chengdu,China)。擴增循環參數為:94℃,30s;60℃,15s;最後在72℃延伸15s。
3、多重PCR
多對引物被混合在同一個PCR反應中進行。最後的反應體系體積為40μL,其中包含PCR Master Buffer(100mM TRIS-HCl,15mM MgCl2,pH8.0),dNTP(200mM)、1.5units TaqPolyemrase(TsingKe Biotech co.,Chengdu,China)、通用正向引物的和物種特異的反向引物(最終的濃度變化範圍是375nM至938nM,見表1)、無菌水和1μL的擴增後的線粒體DNA。PCR的循環參數為:先在如下的循環參數下執行33個循環:95℃,30s、60℃,15s、72℃,15s,然後在72℃延伸3minC(Eppendorf,Germany)。多重PCR的反應條件由Minitab 16的一般線性模型進行優化(Minitab co.,Pennsylvania,USA)。所有的樣品均用3%的瓊脂糖凝膠電泳進行分析,並用Agilent DNA1000生物分析儀進行分析(Agilent Technologies co.,California,USA)。
4、重複性評估
為評估多重PCR結果中引物對內部和相互之間的作用和精確性,我們使用了9個位點特異的引物,5種動物((黃牛(Boridae),犛牛(Boridae),狗(Canidae),豬(Suidae),和雞(Plasianidae))進行了2輪3重複的PCR實驗。用Agilent DNA 1000對PCR產物產量進行評估,並用CV%進行重複性的計算。
5、檢測的低限的確定
因為觀察到鴨肌肉樣品(Anatidae)在凝膠電泳的實驗結果中只有微弱的信號,我們對線粒體DNA模板含量的高低對多重PCR的影響進行了研究。根據我們在實際提取DNA提取過程中所獲得的DNA濃度情況,檢測中我們使用了60ng,6ng,600pg,60ng,和6pg的DNA總量來進行檢測實驗。每份DNA均用REPLI-g線粒體DNA試劑盒(Qiagen,Hilden,Germany)對樣品的線粒體DNA進行了擴增。以此檢測模板對實驗結果的影響。
6、混合樣品檢測
作為跨物種和物種群體組內的混合樣品,研究中製備了含100-%,90-%,80-%,50-%,20-%,10-%犛牛(Boridae)和雞(Plasianidae)樣品的混合物。此外,實驗中也製備了犛牛(Boridae)和黃牛(Boridae)同一物種動物組內的典型的物種混合樣品來進行檢測。
7、特異性檢測
為了檢測多重反應的精確性,實驗準備了9種動物72種組織的盲測樣品來對設計引物和多重PCR的特異性進行檢測。
二、檢測結果
1、多重PCR的結果
在多重PCR中,每個物種均被設計為產生特定條帶的PCR片段,並且每個PCR片段都能與其它8個物種產生的PCR片段成功區分(圖1)。
2、再現性、最低限度、混合物和特異性的檢測結果如下表2所示:
表2再現性、最低限度、混合物和特異性的檢測結果
採用Agilent DNA 1000晶片檢測實驗中三重複的數量來評估組間和組內的重複性。再現性結果如表2所示,特定樣品的PCR產物量在同一批次略高於不同的實驗重複,並且每個PCR產量有不同的重複性。此外,當我們評估Agilent DNA 1000晶片(R&R,Minitab 2016)的標準差時,標準差為19.6%。因此,PCR產物的變異包含在測定系統的變異之中。實驗結果說明,本發明方法的重複性好。
由於多重PCR中需要檢測最低限度的信號,我們用鴨的肌肉樣品(60ngto 6pg)對檢測的低限進行了測定。檢測最低限度的結果如表2所示,本發明方法在基因組DNA為6pg的情況下,仍能準確檢測,說明本發明方法的檢測限度低,靈敏度高。
為檢測混合樣品,我們用黃牛和犛牛的線粒體DNA進行任意混合作為一個實驗動物組來擴增全線粒體DNA。同時將犛牛和雞的樣品按不同的比例(1:9,2:8,5:5,8:2,9:1)混合作為不同物種混合的另一組樣品組。兩個相近物種的混合樣品代表了市場上的假冒商品(經常報導的作假例子)。結果兩個物種可以用本發明方法的多重PCR產物大小順利分開(圖2,表2)。並且PCR產物的產量也依賴於樣品模板DNA的混合比例。在犛牛和雞線粒體DNA混合的例子中,犛牛PCR產物產量甚至沒有達到最低的線粒體DNA濃度(犛牛:雞=10:90),但是雞的PCR產物產量增加到了給定的線粒體DNA量。研究結果表明,根據模板濃度的不同,多重PCR引物競爭將產生不同的產物模式。
為檢測本研究方法的實際擴增效果和準確度,我們對從中國西南部採集到的72份動物肉樣進行了檢測。結果如表2和圖3所示,本發明方法可以準確檢測71個樣品,樣品準確度幾乎達到100%。
三、討論
在實際的市場上,有許多混合的肉類產品。因此,本研究建立的肉類鑑定檢測方法的可靠性仍需要使用混合樣品進行確認。犛牛和黃牛均屬於牛科,從遺傳角度看區分它們是比較困難的。因此,用我們建立的方法對犛牛和黃牛的混合樣品是進行區分是最好例子。由於經濟利益驅使,市場上它們的混合產品也較多。我們的檢測結果表明,我們的檢測方法能夠清晰的區分犛牛和黃牛樣品。此外,即使在檢測反應中只加入少量的線粒體DNA(例如只加入我們研究方法中10%線粒體DNA,見圖2和表2),該方法也能將之與其它物種順利區分。與陳研究小組(2010)建立的混合PCR方法在混合樣品20%的檢測底限的結果相比,本研究建立的檢測低限更低(10%)。當我們檢測不同家族混合樣品時(例如,以犛牛和雞混合製作樣品),本方法也能很好的區分彼此。
同時本發明用真實的肉類產品對我們方法的可靠性進行檢測,實際使用了從市場上採集的9個物種的72份真實肉樣進行了檢測。結果有71份樣品完全吻合它們的檢測標誌(表2和圖3)。這些結果表明,我們的檢測方法優於Naue建立的HRM分析方法的特異性(28個肉樣中的24個,Naue et al,2014)。
四、結論
本發明PCR檢測方法可以對包括犛牛等青藏高原物種進行區分,該方法的重現性好、檢測限低、特異性好,可以對混合樣品進行檢測。
綜上,本發明引物、試劑盒和PCR方法可以特異擴增各種不同的肉類,特異性好,而且靈敏度高,重現性好,為現有肉類檢測提供了一種權威的認證方法,可用於在市場上消除虛假犛牛肉和肉類產品,市場應用前景優良。
SEQUENCE LISTING
西南民族大學
一種肉類檢測試劑盒和方法
GY223-16P1532
22
PatentIn version 3.5
1
25
DNA
P1-1F
1
gtgacaaaaa ttaagccata aacga 25
2
22
DNA
1_Ca_P1-1R
2
tcatggcttt ttacagcttg gt 22
3
21
DNA
P2-2F
3
gataccccac tatgcttagc c 21
4
22
DNA
2-Ya-P2-1R
4
atttcataat aactttcgtg ca 22
5
24
DNA
P1-1F
5
gtgacaaaaa ttaagccata aacg 24
6
25
DNA
4_Bu_P1-2R
6
gctgttatag agtcactttc gttgc 25
7
24
DNA
P1-1F
7
gtgacaaaaa ttaagccata aacg 24
8
22
DNA
5_Go_P1-2R
8
gatttaccaa ccctggtcaa ca 22
9
21
DNA
P2-2F
9
gataccccac tatgcttagc c 21
10
21
DNA
7_Do_P2-1R
10
acattcgtga aatgttcttg g 21
11
24
DNA
P1-1F
11
gtgacaaaaa ttaagccata aacg 24
12
20
DNA
8_Pig_P1_1R
12
aacactcttt acgccgtgga 20
13
21
DNA
P2-2F
13
gataccccac tatgcttagc c 21
14
20
DNA
9-Ra-P2-1R
14
acctaacgtt tttacggtgg 20
15
21
DNA
P2-2F
15
gataccccac tatgcttagc c 21
16
21
DNA
10-Ch-P3-1R
16
gctattgagc tcactgttgt t 21
17
21
DNA
P2-2F
17
gataccccac tatgcttagc c 21
18
17
DNA
11-Du-P3-2R
18
aaggggcggt tgggttg 17
19
25
DNA
P1-1F
19
gtgacaaaaa ttaagccata aacga 25
20
21
DNA
P1-1R
20
agggctaagc atagtggggt a 21
21
21
DNA
P2-2F
21
gataccccac tatgcttagc c 21
22
23
DNA
P2-2R
22
gaatctattt gaggagggtg acg 23
