應用三代測序技術檢測染色體端粒DNA全長的方法與流程
2023-05-28 10:41:01 3
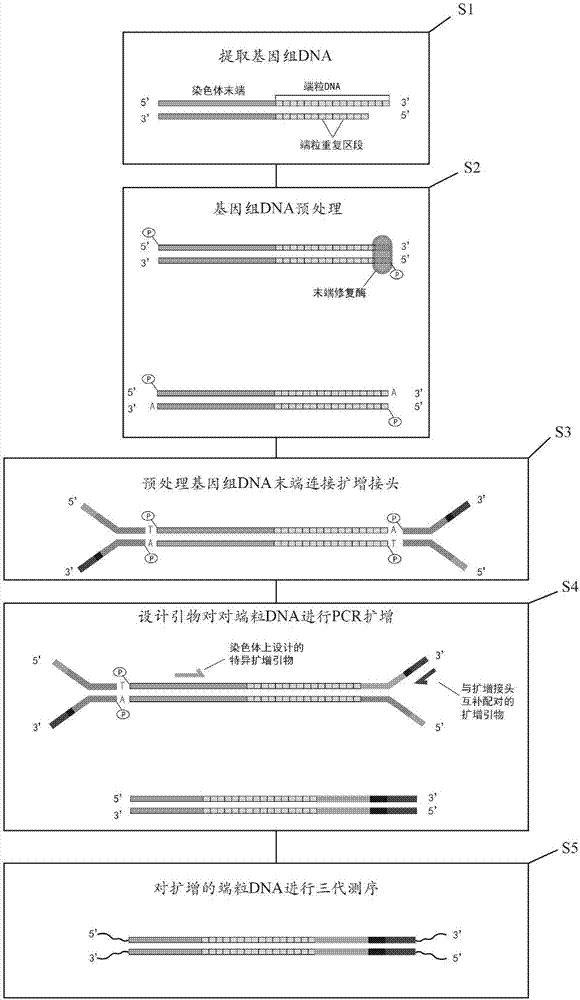
本發明涉及分子生物學領域,特別涉及應用三代測序技術檢測染色體端粒dna全長的方法。
背景技術:
:端粒是位於真核細胞線性染色體末端的一小段dna-蛋白質複合體,其作用是保護染色體dna的末端,避免染色體dna的降解、末端融合以及非正常重組等。在哺乳動物細胞中,端粒dna由ttaggg序列和aatccc序列重複串聯組成。由於dna的末端複製問題,細胞的每次分裂都伴隨著端粒dna一小段序列的丟失。細胞的每次分裂都會伴隨著染色體末端端粒的縮短,細胞也因此逐漸老化、直至喪失增殖能力而死亡。端粒長度的變化與多種疾病密切相關,如癌症、早衰症候群等。因此測量端粒長度能提供與疾病相關的重要信息。目前測量端粒長度主要是基於qpcr的方法,對重複區段進行擴增,端粒dna的片段越長,重複區段的拷貝數就多。但是這種方法只能檢測所有染色體端粒重複區段長度的大概平均值,無法精確檢測單獨每一條染色體的端粒長度。而且由於染色體dna其他部分也存在類似端粒dna重複序列的結構,這部分序列也會影響qpcr對端粒長度的檢測。從實驗操作上來說,由於qpcr反應很容易受到其他條件影響,基於qpcr測量方法的定量結果穩定性不好,如果結果ct值相差1,會導致端粒長度測量結果增加1倍。因此,研發新的、更準確的測量端粒dna長度的方法是本領域技術人員亟需解決的技術問題。技術實現要素:有鑑於此,本發明提供的應用三代測序技術檢測染色體端粒dna全長的方法,能有效解決當前不能獲得端粒dna全長和準確測量單條染色體端粒dna的技術缺陷。為了實現上述發明目的,本發明提供以下技術方案:本發明公開了染色體端粒dna的擴增方法,包括下列步驟:1)提取基因組dna;2)將步驟1)的基因組dna進行預處理;3)將步驟2)獲得的預處理後的基因組dna兩端的末端連接擴增接頭,得到帶有擴增接頭的基因組dna;4)分別對步驟3)的帶有擴增接頭的基因組dna序列和擴增接頭序列設計引物對,對端粒dna進行pcr擴增。作為優選,所述步驟2)的基因組dna進行預處理為將所述基因組dna的兩端進行末端修復。作為優選,所述步驟2)基因組dna兩端進行末端修復為將基因組dna片段的末端補平,然後對基因組dna的5』端進行磷酸化,以及將基因組dna片段的3』端添加腺嘌呤da。更為優選,所述腺嘌呤da為脫氧腺嘌呤da。作為優選,所述步驟3)的擴增接頭還包括標籤序列。其中,所述步驟3)的擴增接頭還包括標籤序列,具體為所述擴增接頭與所述標籤序列重組後的新型擴增接頭。所述新型擴增接頭除了擴增接頭外還包括標籤序列。其中,所述擴增接頭直接與步驟2)的預處理後的基因組dna連接,或所述擴增接頭與所述標籤序列重組後的新型擴增接頭與步驟2)的預處理後的基因組dna連接。其中,所述步驟4)擴增得到的端粒dna產物的序列包括端粒dna序列和標籤序列。作為優選,所述步驟3)的擴增接頭的標籤序列為與人類基因組dna比對的evalue大於1的序列,不會對後續分析造成幹擾。其中,所述標籤序列用於區分不同人來源的染色體端粒dna。作為優選,為了能應用於大樣本量的項目,所述步驟3)的擴增接頭的標籤序列為16個bp。作為優選,所述步驟3)還包括將得到的帶有擴增接頭的基因組dna進行純化,以便獲得高質量的帶有擴增接頭的基因組dna。作為優選,所述擴增接頭為y型接頭、環狀接頭或線性接頭中的一種。更為優選,所述擴增接頭為y型接頭。其中,所述y型接頭為形狀像y,根部互補,其餘部分不互補的dna序列。本發明還提供了一種染色體端粒dna的擴增試劑盒,包括基因組dna提取體系、基因組dna預處理體系、擴增接頭、擴增接頭與基因組dna連接體系和端粒dna的pcr體系。其中,所述基因組dna提取體系用於提取人體基因組dna;所述基因組dna預處理體系用於對基因組dna的末端進行補平,5』端磷酸化和3』端添加腺嘌呤da;所述擴增接頭與基因組dna連接體系用於將所述擴增接頭連接到所述預處理後的基因組dna末端;所述端粒dna的pcr體系用於擴增基因組dna5』端和擴增基因組dna3』端的端粒片段。作為優選,所述基因組dna預處理體系包括基因組dna的末端補平體系、基因組dna的末端磷酸化體系和基因組dna的末端3』端進行添加腺嘌呤da體系。作為優選,所述基因組dna的末端補齊體系為基因組dna的平末端修復體系;所述基因組dna的平末端修復體系包括t4dnapolymerase或/和dna聚合酶i大片段(klenow片段)。作為優選,所述基因組dna的末端磷酸化體系包括t4多聚核苷酸激酶。作為優選,所述基因組dna片段的3』端進行添加腺嘌呤da體系包括klenow(exon-)。作為優選,所述擴增接頭與基因組dna連接體系包括連接酶。所述連接酶為t4dna連接酶。作為優選,所述擴增接頭如seqidno.1~seqidno.31所示。其中,所述基因組dna預處理可以在一個反應中完成,或多個反應中完成。作為優選,所述端粒dna的pcr體系的引物對為基因組dna的5』端非端粒區的引物和所述擴增接頭的引物用於擴增基因組dna的5』端端粒,或基因組dna的3』端非端粒區的引物和所述擴增接頭的引物用於擴增基因組dna的3』端端粒。其中,所述基因組dna的5』端非端粒區的引物為5』基因組dna的近端粒區的非端粒區引物;所述基因組dna的3』端非端粒區的引物為3』基因組dna的近端粒區的非端粒區引物。其中,所述端粒dna的pcr體系的引物對包括以下的可能(從5』到3』的單鏈dna):1、擴增接頭序列的引物和近5』端的端粒的非端粒區基因組dna反向互補引物;2、近3』端的端粒的非端粒區基因組dna引物和擴增接頭序列反向互補引物。其中,與所述擴增接頭互補配對的引物和所述近3』端的端粒的非端粒區基因組dna引物,或近5』端的端粒的非端粒區基因組dna反向互補引物和擴增接頭上的引物的具體長度不做限定。作為優選,所述端粒dna的pcr體系包括預處理後帶有擴增接頭的基因組dna、擴增端粒dna的pcr引物對、pcr工具酶、dntp和pcr反應緩衝液。所述引物對由接頭通用pcr引物和端粒dna特異性引物構成,所述通用pcr引物如seqidno.32所示,端粒dna特異性引物如seqidno.33~seqidno.35所示。本發明還公開了應用三代測序技術檢測染色體端粒dna全長的方法,包括下列步驟:1))提取基因組dna片段;2))將步驟1))的基因組dna片段進行預處理;3))步驟2))獲得的預處理後的基因組dna片段兩端的末端添加擴增接頭,得到帶有擴增接頭的基因組dna片段;4))分別對步驟3))的帶有擴增接頭的基因組dna片段的基因組dna序列和擴增接頭序列設計引物對,對端粒dna進行pcr擴增;5))將步驟4))的pcr擴增的端粒dna構建三代測序文庫後,將所述三代測序文庫上機測序,得到測序數據。本發明還提供了應用三代測序技術檢測染色體端粒dna全長的試劑盒,包括基因組dna提取體系、基因組dna預處理體系、擴增接頭、擴增接頭與基因組dna連接體系、端粒dna的pcr體系和三代測序文庫構建體系。其中,所述三代測序文庫構建體系用於構建擴增的端粒dna進行三代測序文庫,綜上所述,本發明公開了染色體端粒dna的擴增方法,該方法能特異性的擴增染色體端粒dna的全長,而且,還能通過標籤序列區分不同人的染色體端粒dna,採用不同引物還能擴增不同染色體的端粒dna,而且該擴增方法步驟簡單,特異性強。本發明還公開的應用三代測序技術檢測染色體端粒dna全長的方法能同時測量不同人、不同染色體的端粒dna,所擴增的端粒dna全長片段使用3代測序方法能準確測量端粒dna序列長度。先富集基因組dna的端粒後在測序端粒dna序列,大大提高了測序端粒dna的準確率和精度,而且還能降低成本。本發明公開的檢測染色體端粒dna全長的方法能達到單鹼基解析度,彌補了特異性地準確測量單條染色體完整端粒dna技術的空白。附圖說明為了更清楚地說明本發明實施例或現有技術中的技術方案,下面將對實施例或現有技術描述中所需要使用的附圖作簡單地介紹。圖1示應用三代測序技術檢測染色體端粒dna全長的方法介紹圖;圖2示應用三代測序技術檢測染色體端粒dna全長的方法原理步驟說明圖;圖3示應用三代測序技術檢測染色體端粒dna全長的方法的膠圖,其中序號1為基因組dna膠圖(m為marker,1、2、3欄為樣品1、樣品2、樣品3的基因組dna)、序號2為預處理後基因組dna連接擴增接頭的膠圖(m為marker,1、2、3欄為樣品1、2、3中基因組dna連接擴增接頭後的dna)、序號3為端粒dna(m為marker,1、2、3欄為一號染色體、二號染色體和七號染色體端粒dna片段);圖4示nanoporeminion測序的gc含量分析,藍色線是總的數據集,紅色線是篩選過濾後的端粒dna的reads;圖5示nanoporeminion測序的reads的讀長分析,藍色線是總的數據集,紅色線是篩選過濾後的端粒dna的reads;圖6示端粒dna中ttaggg或者其互補序列aatccc重複出現在reads中的次數;圖7示本發明的方法與對比例方法測量端粒dna長度結果比較圖,其中橫坐標chr1、chr2和chr7為300例樣品中一號染色體、二號染色體和七號染色體的端粒dna全長分布;對比例方法為通過qpcr方法間接測量300例樣品所有染色體端粒dna的平均長度分布。其中,chr1為一號染色體,chr2為二號染色體,chr7為七號染色體。具體實施方式本發明公開了應用三代測序技術檢測染色體端粒dna全長的方法,本領域技術人員可以借鑑本文內容,適當改進工藝參數實現。特別需要指出的是,所有類似的替換和改動對本領域技術人員來說是顯而易見的,它們都被視為包括在本發明。本發明的方法及應用已經通過較佳實施例進行了描述,相關人員明顯能在不脫離本
發明內容、精神和範圍內對本文所述的方法和應用進行改動或適當變更與組合,來實現和應用本發明技術。本發明提供的應用三代測序技術檢測染色體端粒dna全長的方法,其中所用原料及試劑均可由市場購得。實施例1對中青年人和老年人的端粒dna進行測序,具體方法如下:選取150個在18~45歲的中青年人,男女各75個;選取150個60~90歲的老年人,男女各75個。提取150個中青年人和150個老年人組的基因組dna,具體步驟如下:1、提取上述人群中血液的dna,做好標記;2、使用試劑盒illustradnaextractionkitphytopure(genucleonrpn-8510)提取高質量大片段基因組dna。對獲取的300例樣品的基因組dna進行瓊脂糖凝膠電泳檢測基因組dna,如圖1所示,1、2和3欄為樣品1、2和3基因組dna電泳圖,基因組dna片段基本分布在15k以上。實施例2對實施例1提取的基因組dna處理,具體步驟如下:1、測定實施例1基因組dna濃度,對基因組dna片段進行末端修復,將基因組dna的末端補平、磷酸化,採用如下基因組dna的末端補平、磷酸化體系(100μl)進行基因組dna的末端補平、磷酸化:將以上基因組dna的末端補平、磷酸化體系混合後,22℃反應20min,柱式法純化,50-100μlte洗脫,得到混合物1。2、對步驟1的混合物1獲得的基因組dna片段的3』端進行添加da,採用如下加da體系(100μl)進行:將以上加da體系完全混合後,置於37℃反應20min,柱式法純化後,50-100ulte洗脫,得到基因組dna預處理後的基因組dna片段。實施例3對實施例2經過基因組dna末端修復、5』端磷酸化和3』端添加腺嘌呤da的基因組dna的末端連接帶有標籤序列的擴增接頭,其中,標籤序列設於擴增接頭中,在本實施例中,標籤序列的長度為16bp,使得擴增接頭區分不同人的端粒dna。擴增接頭序列可以為y型、圓環型、線狀型,本實施例使用y型擴增接頭,實施例1中150個中青年人和150個老年人總共有300個樣品,分別設計300個標籤序列製備的y型擴增接頭區分不同樣品,本實施例只列舉30個y型擴增接頭。1、y型擴增接頭製備方法如下:y型擴增接頭由兩條引物退火連接而成,y型擴增接頭的引物序列如seqidno.1~seqidno.31所示,引物組合方式如表1所示(擴增接頭1由seqidno.1與seqidno.2退火連接而成;擴增接頭2由seqidno.1與seqidno.3退火連接而成;擴增接頭3由seqidno.1與seqidno.4退火連接而成,以此類推),其中,seqidno.2~seqidno.31的引物5』端需要進行磷酸化修飾以提高連接效率,引物使用引物稀釋液(10mmtris-hcl,ph7.5)稀釋至終濃度為100μm。分別將兩個引物取30μl等體積混合到200μlpcr管中,放置在pcr熱循環儀中,通過以下程序參數形成y型接頭:5minutes95℃;5minutes72℃;-0.1℃/sdropdownto20℃,保持5minutes;holdat4℃。表1y型接頭統計表2、y型擴增接頭與基因組dna片段連接方法,具體步驟如下:y型擴增接頭使用引物稀釋液(10mmtris-hcl,ph7.5)稀釋至終濃度為1.0μm。使用ultratmligationmodule(nebe7445l)試劑盒進行接頭連接,按照試劑盒說明書製備體系;使用移液槍輕微吹打均勻反應體系,pcr管短暫離心,把反應體系液體收集到管底,並放入pcr熱循環儀中,開啟熱蓋45℃,20℃反應15minutes。圖中2表示基因組dna與y型擴增接頭連接後的產物的電泳圖,同樣取了樣品1、2、和3作為電泳圖展示。實施例4對實施例3得到的帶有擴增接頭的基因組dna片段進行純化,具體步驟如下:使用agencourtampurexp(beckmancoulter,a63881)核酸純化磁珠純化回收接頭連接產物。步驟:取30μlampurexpbeads,放置到新的pcr管中,室溫平衡30minutes以上,加入到上述接頭連接步驟反應體系中,使用移液槍輕微吹打均勻,室溫放置10minutes。pcr管放到磁力架上吸附磁珠,等待5minutes後,使用移液槍移去上清,並加入140μl新鮮配置的80%乙醇(使用ddh2o配置),蓋上管蓋並保持在磁力架上,室溫放置10minutes。使用移液槍移去上清。重複用140μl新鮮配置的80%乙醇清洗磁珠。移去所有乙醇後,打開管蓋,室溫晾乾5minutes,揮發乙醇。pcr管從磁力架上拿下來,加入30μl洗脫液(10mmtris-hcl,ph8.0)吹打均勻,室溫放置10minutes,重新放在磁力架上吸附磁珠,包含純化的接頭連接產物的上清液體移到新的pcr管中,-20℃保存。實施例5分別在實施例4所述的基因組dna和所述的擴增接頭序列設計特異性引物對端粒dna進行pcr擴增,具體步驟如下:所述端粒dna的pcr體系包括基因組dna的5』端非端粒區引物和所述擴增接頭的引物用於擴增基因組dna的5』端端粒;或,基因組dna的3』端非端粒區引物和所述擴增接頭的引物用於擴增基因組dna的3』端端粒。具體來說,在5』到3』方向,pcr擴增引物為,所述擴增接頭的引物和與近5』端端粒區的基因組dna反向互補的引物,或在5』到3』方向,近3』端端粒區的基因組dna引物和與所述擴增接頭反向互補的引物。pcr引物對如seqidno.32~seqidno.35所示,兩個引物等量混合,使用引物稀釋液(10mmtris-hcl,ph7.5)稀釋至終濃度10μm,組成primermix。pcr引物對如表2所示。表2pcr引物對使用gcgenomiclapolymerasemix試劑盒(takara,codeno.639153)擴增端粒dna長片段。根據試劑盒說明配置pcr擴增反應體系組分。使用移液槍輕微吹打均勻反應體系,短暫離心,把擴增反應體系液體收集到管底,並放入pcr熱循環儀中,開啟熱蓋105℃,擴增反應程序參數如下:s1:95℃for5minutes;s2:95℃for30sec;s3:60℃for30sec;s4:72℃for15minutes;重複s2~s4:30cycles;s5:72℃for15minutes;s6:holdat4℃。pcr擴增產物加入75μlddh2o擴充至100μl,然後加入40ulampurexpbeads,如實施例4操作步驟,純化回收擴增產物,最後使用30μl洗脫緩衝液洗脫,獲得高質量的帶有擴增接頭的基因組dna,將帶有擴增接頭的基因組dna進行瓊脂糖凝膠電泳檢測,結果如圖3所示。圖中3是通過染色體1、染色體2和染色體7近染色體端的特異性引物和接頭引物,分別特異性擴增染色體1,染色體2和染色體7的全長端粒dna片段。儘管有少許非特異性,但是本發明方法能夠非常高效和特異地擴增染色體端粒dna全長片段。實施例6nanopore測序文庫構建建庫試劑盒用nanopore的試劑盒,1dsequencingkit(sqk-rad002rapidsequencingkit)將實施例5的帶有擴增接頭的基因組dna產物按照nanopore試劑盒的說明書進行文庫構建。上機測序及數據分析,具體步驟如下:1、按照nanoporeminionsequencer的要求上機測序:上樣到r9.4/flo-min106flowcell中,經過minionmk1b測序儀器檢測。2、按照測序數據分析pipeline將測序數據轉化為fastq格式;3、數據分析:a、過濾不包含與所述擴增接頭互補配對的引物和基因組dna上的引物的reads和,與所述基因組dna互補配對的引物和擴增接頭上的引物的reads;b、過濾不包含重複ttaggg和ccctaarepeats的序列;c、統計過濾後reads的長度分布;d、輸出分析結果。其中,fastaq數據中的篩選符合條件的reads,篩選條件為同時有與所述擴增接頭互補配對的引物和基因組dna上的引物的reads和,與所述基因組dna互補配對的引物和擴增接頭上的引物的reads,及同時序列中具有ttaggg或aatccc重複序列的reads。通過接頭序列中的barcode,區分不同人的樣品。另外通過graphmap0.3.1軟體比對將這些reads對比到人類近染色體端序列,區分不同染色體的reads,然後計算相應染色體reads的長度。並做如下的分析。表3nanoporeminion測序總體情況sequence通過質檢的端粒dnareadsreads3104125857totalbases269823055197780299readsaveragelength(bp)86927649其中,nanoporeminion測序總體情況對300例樣品的測序總體情況如表3所示,總共測出310401條序列,其中端粒dnareads數量為258564條,其中端粒dna的平均長度為8325bp。圖4至圖7為nanoporeminion測序的數據分析,圖4示全部reads中gc含量統計圖,其中,藍色線是總的數據集,紅色線是篩選過濾後的端粒dna測序reads,篩選過濾的條件為:reads兩端分別包含與所述擴增接頭互補配對的引物和基因組dna上的引物和,與所述基因組dna互補配對的引物和擴增接頭上的引物,另外reads中包含ttaggg或ccctaa重複序列。本發明方法得到的端粒dnareads中gc含量約為62.5%,符合端粒dna的鹼基組成。本發明檢測端粒dna長度的結果如表4所示。表4端粒dna長度的結果統計對實施例6中圖7和表4的說明如下:中青年組chr1端粒dna長度的平均值為9267bp,老年人組chr1端粒dna長度的平均值為8913bp;中青年組chr2端粒dna長度的平均值為9184bp,老年組chr2端粒dna長度的平均值為7638bp;中青年組chr3端粒dna長度的平均值為6925bp,老年組chr3端粒dna長度的平均值為4895bp。在chr1、chr2及chr3的端粒dna長度測序結果中,老年組的端粒dna平均長度均比中年組要短,說明老年組的端粒整體上相比於中青年組的要顯著縮短,p<0.05。中青年組及老年組chr1、chr2及chr7端粒測序結果的箱線圖見圖7。對比例中圖7和表4的說明的說明如下:對比例qpcr方法測得的中青年組的端粒dna的平均長度為8326bp,對比例測得的老年組的端粒平均長度為7356bp,老年組的端粒長度也相比於中青年組要短,中青年組及老年組qpcr檢測端粒dna平均長度結果的箱線圖同見圖7。圖5為reads的讀長分析,其中,藍色線是總的數據集,紅色線是篩選過濾後的端粒dna測序reads,篩選過濾的條件如上述,圖5所示,端粒dna的reads中包含部分近端粒區序列(約1~2kbp),可以發現,端粒dna測序reads讀長符合已知染色體端粒長度分布範圍(4-10kbp)。圖6為端粒dna的reads中ttaggg或者其互補序列aatccc重複出現在總reads中的次數分布圖。從圖6中可以看到,測序的端粒dnareads片段中,存在大量的ttaggg/aatccc重複序列,符合已知端粒dna序列的組成。經過統計,測序數據中符合端粒dna條件的reads中,ttaggg/aatccc重複序列平均重複出現次數1200次。對比例採用telomeremeasurementbyquantitativepcr(richardm.cawthon,nucleicacidsresearch,2002)分析端粒dna的方法,該方法是採用了t/s比率的比率,即t/s比率可以得出端粒的相對長度,而t/s比率與端粒長度成正比關係。t/s計算公式如下:t/s=[2ct(telomeres)/2ct(36b4)]=2-δct。端粒長度可以通過標準曲線y=1910.5x+4157計算,其中x為t/s比率,y為端粒dna的平均長度。使用該方法計算中青年人組和老年人組全部染色體的端粒的平均長度,結果如圖7中對比例方法所示。圖7為本發明方法與對比例方法測量端粒dna長度比較圖。圖7的chr1、chr2和chr7為通過標籤序列可以區分不同人的染色體序列,篩選的端粒dnareads,使用graphmap軟體與人的一號染色體、二號染色體和七號染色體的近端粒區域比對,通過比對結果區分端粒dnareads屬於哪條染色體,同時該reads長度減去reads中部分近端粒的染色體dna長度,獲得該染色體端粒的長度,統計每個人一號染色體、二號染色體和七號染色體端粒dna的長度。而對比例的測量端粒dna的方法只能測量基因組上所有染色體端粒dna的平均長度。分別對中青年人和老年人組一號染色體、二號染色體和七號染色體的端粒長度做分布圖,發現不同染色體中,端粒dna長度具有差異。另外同時發現,中青年人組中一號染色體、二號染色體和七號染色體的端粒長度均大於老年人組,符合以往研究。對比例方法中同時也驗證了中青年人組端粒dna長度大於老年人組,但是由於傳統方法只能檢測基因組所有染色體端粒的總體長度水平,無法精確測量每個染色體的端粒長度,所以在檢測結果上,置信區間的分布較本發明的大,不利於準確比較端粒的長度。綜上所述,本發明公開的應用三代測序技術檢測染色體端粒dna全長的方法能特異性的擴增不同染色體的端粒dna全長,所擴增全長的端粒dna片段使用3代測序方法準確測量其片段序列長度,準確率和精度都超過傳統的方法,本發明公開的檢測染色體端粒dna全長的方法精度達到1bp。以上所述僅是本發明的優選實施方式,應當指出,對於本
技術領域:
的普通技術人員來說,在不脫離本發明原理的前提下,還可以做出若干改進和潤飾,這些改進和潤飾也應視為本發明的保護範圍。sequencelisting廣州普麥健康諮詢有限公司應用三代測序技術檢測染色體端粒dna全長的方法mp171056135patentinversion3.5150dna人工序列1ggcgacctccgagatctacactctttacctacacgagcctcctacgtact50263dna人工序列2gtacgtaggaggccacgtcttaccgacagtgaccttcgtatgtagtcttctgcttccgat60cga63363dna人工序列3gtacgtaggaggccacgtctcggattccaaggtactcgtatgtagtcttctgcttccgat60cga63463dna人工序列4gtacgtaggaggccacgtctcctcatggtcagtcatcgtatgtagtcttctgcttccgat60cga63563dna人工序列5gtacgtaggaggccacgtccgaatacgtctaggagtcgtatgtagtcttctgcttccgat60cga63663dna人工序列6gtacgtaggaggccacgtccgcaactacgtatctctcgtatgtagtcttctgcttccgat60cga63763dna人工序列7gtacgtaggaggccacgtcgtacacctcggcattatcgtatgtagtcttctgcttccgat60cga63863dna人工序列8gtacgtaggaggccacgtctcgtctgaagcctatctcgtatgtagtcttctgcttccgat60cga63963dna人工序列9gtacgtaggaggccacgtcctgtaccaagaccttgtcgtatgtagtcttctgcttccgat60cga631063dna人工序列10gtacgtaggaggccacgtcaggatagcccttccattcgtatgtagtcttctgcttccgat60cga631163dna人工序列11gtacgtaggaggccacgtcgtccttgatgcgtaactcgtatgtagtcttctgcttccgat60cga631263dna人工序列12gtacgtaggaggccacgtccacagactttacgtgctcgtatgtagtcttctgcttccgat60cga631363dna人工序列13gtacgtaggaggccacgtcccaaggttcacgcaattcgtatgtagtcttctgcttccgat60cga631463dna人工序列14gtacgtaggaggccacgtcttccaggttcatctcctcgtatgtagtcttctgcttccgat60cga631563dna人工序列15gtacgtaggaggccacgtcgagagtacgtcgttactcgtatgtagtcttctgcttccgat60cga631663dna人工序列16gtacgtaggaggccacgtcggaggaataggaacactcgtatgtagtcttctgcttccgat60cga631763dna人工序列17gtacgtaggaggccacgtccagtgcttcttgctagtcgtatgtagtcttctgcttccgat60cga631863dna人工序列18gtacgtaggaggccacgtctggaagcaagctaagctcgtatgtagtcttctgcttccgat60cga631963dna人工序列19gtacgtaggaggccacgtcgaacggttggaatgtctcgtatgtagtcttctgcttccgat60cga632063dna人工序列20gtacgtaggaggccacgtctacggtctccagtatctcgtatgtagtcttctgcttccgat60cga632163dna人工序列21gtacgtaggaggccacgtctctacgcagtaaccgatcgtatgtagtcttctgcttccgat60cga632263dna人工序列22gtacgtaggaggccacgtcgacgtcatcttacagctcgtatgtagtcttctgcttccgat60cga632363dna人工序列23gtacgtaggaggccacgtctccattgcagcgagattcgtatgtagtcttctgcttccgat60cga632463dna人工序列24gtacgtaggaggccacgtccaatagccaagacacctcgtatgtagtcttctgcttccgat60cga632563dna人工序列25gtacgtaggaggccacgtcccagttgatggtgaagtcgtatgtagtcttctgcttccgat60cga632663dna人工序列26gtacgtaggaggccacgtcaagaccgtttgcgagatcgtatgtagtcttctgcttccgat60cga632763dna人工序列27gtacgtaggaggccacgtcgcaattccgaatccgttcgtatgtagtcttctgcttccgat60cga632863dna人工序列28gtacgtaggaggccacgtcccgcttaatacctgcatcgtatgtagtcttctgcttccgat60cga632963dna人工序列29gtacgtaggaggccacgtcgtcgattgtatggcactcgtatgtagtcttctgcttccgat60cga633063dna人工序列30gtacgtaggaggccacgtcctcgaacacaactccatcgtatgtagtcttctgcttccgat60cga633163dna人工序列31gtacgtaggaggccacgtctgcatcgtggacatcatcgtatgtagtcttctgcttccgat60cga633221dna人工序列32cgatcggaagcagaagactac213320dna人工序列33caactccgccgttgcaaagg203421dna人工序列34cgtatcccacacaccacaccc213520dna人工序列35cagacagtgcacaaagccac20當前第1頁12
