一種高效的酵母菌染色體融合方法與流程
2023-05-24 09:56:56 1
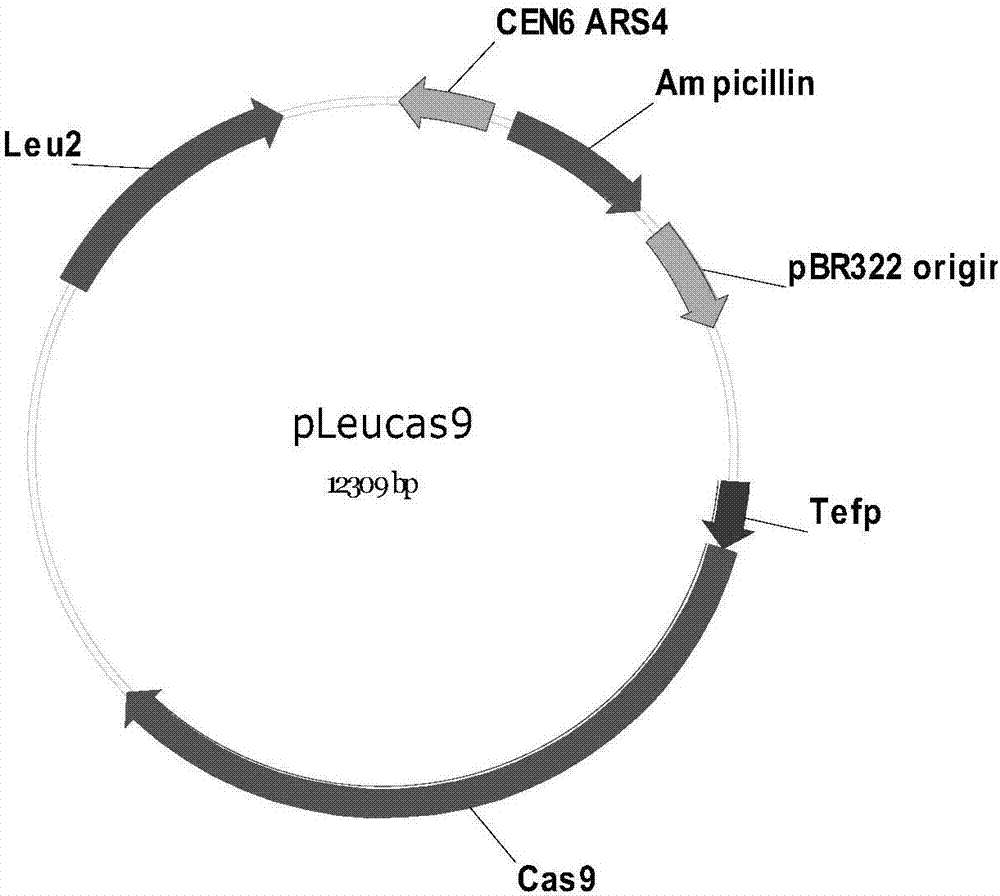
本發明涉及微生物合成生物學、基因組工程和分子生物學領域,具體涉及一種高效的酵母菌染色體融合方法。
背景技術:
:研究表明,酵母可以作為珍稀藥物前體青蒿酸、紫杉烯、氫化可的松和新能源丁醇的異源表達宿主。還因其高效的同源重組能力,可以作為dna大片段克隆及其他微生物基因組組裝和改造的宿主菌,特別是對於實驗室條件下難培養、缺乏有效遺傳操作手段的菌株。釀酒酵母(saccharomycescerevisiae)是基礎研究最透徹、工業應用最廣泛的模式生物之一。然而,釀酒酵母天然有16條線性染色體,大小在230kbp到1530kbp,造成了在酵母體內克隆的超過200kbp的大片段dna難以和染色體進行分離。這一缺陷限制了釀酒酵母作為異源基因表達和大片段dna克隆、基因組改造宿主的應用。因此,本領域有必要發展快速高效的染色體融合新技術來構建新的基因組簡約化且遺傳穩定的釀酒酵母宿主菌。技術實現要素:本發明的目的在於提供一種高效的酵母菌染色體融合方法。在本發明的第一方面,提供一種對酵母的染色體a和染色體b進行融合的方法,其特徵在於,所述方法包括:將著絲粒敲除組件、染色體融合組件、sgrna靶向切割組件引入到表達cas9蛋白的酵母中,從而獲得染色體a和染色體b發生融合的酵母;其中,所述著絲粒敲除組件用於敲除兩條染色體(選自染色體a和染色體b)之一的著絲粒,其含有該著絲粒兩側同源序列和篩選標記(較佳地,篩選標記位於兩條同源序列之間);所述染色體融合組件用於在兩條染色體兩個端粒附近發生雙交換,刪除端粒且介導染色體融合,其含有染色體a近端粒區的同源序列、染色體b近端粒區的同源序列和篩選標記(較佳地,篩選標記位於兩條同源序列之間);所述sgrna靶向切割組件含有能在要刪除的著絲粒和兩個端粒序列附近進行切割的sgrna。在一個優選例中,所述的cas9表達組件是表達載體,其中以酵母強啟動子驅動cas9基因的表達;較佳地,所述的強啟動子為tef1啟動子。在另一優選例中,所述的cas9表達組件是表達載體,其在酵母中為單拷貝複製、在大腸桿菌中為高拷貝複製;較佳地,所述的表達載體的載體複製區來源於cen6ars4;較佳地,所述的表達載體以leu2作為篩選標記;較佳地,所述的表達載體的載體複製起始位點為pbr322origin(來源於pbr322)。在另一優選例中,所述的cas9表達組件的骨架載體具有seqidno:1所示的核酸序列。在另一優選例中,所述sgrna靶向切割組件是能在酵母中組成型表達sgrna的表達載體;較佳地,其以酵母強啟動子驅動sgrna的表達;較佳地,所述的強啟動子為snr52。在另一優選例中,所述sgrna靶向切割組件是能在酵母中組成型表達sgrna的表達載體,其在酵母中為單拷貝複製、在大腸桿菌中為高拷貝複製;較佳地,所述的表達載體的載體複製區為2micron2origi(來源於2μm質粒);較佳地,所述的表達載體以his3作為酵母表達的篩選標記;以氨苄青黴素抗性基因作為大腸桿菌表達的篩選標記;較佳地,所述的表達載體的載體複製起始位點為pbr322origin(來源於pbr322)。在另一優選例中,所述的sgrna靶向切割組件的骨架載體具有seqidno:2所示的核酸序列。在另一優選例中,在著絲粒敲除組件中,設置一段正向重複序列,其設置於該組件中所述同源序列與篩選標記之間;其序列為染色體上緊挨該著絲粒一側同源序列的序列;較佳地,該正向重複序列的長度為50~500bp(例如,100~300bp;更佳地如150bp,180bp,200bp,250bp)。在另一優選例中,所述染色體融合組件中,設置一段正向重複序列,設置於該組件中所述同源序列與篩選標記之間;其序列為緊挨著其中一條染色體近端粒區同源序列的序列;較佳地,該正向重複序列的長度為50~500bp(例如,100~300bp;更佳地如150bp,180bp,200bp,250bp)。在另一優選例中,通過酵母原生質體轉化方法將著絲粒敲除組件、染色體融合組件、sgrna靶向切割組件、cas9表達組件引入到酵母中。在另一優選例中,將釀酒酵母的染色體chrvi和chri進行融合,其中,所述著絲粒敲除組件用於敲除chrvi著絲粒,並且,所述的著絲粒兩側同源序列分別為左同源臂chrvi:148410-148459、右同源臂chrvi:148725-148774,所述正向重複序列為chrvi:148775-149002;所述染色體融合組件中,兩條染色體的近端粒區的同源序列分別為chrvi:269212-269615和chri:2893-3294,所述正向重複序列為chri:3295-3519;所述sgrna靶向切割組件含有seqidno:3、seqidno:4、seqidno:5(對應於target1~3)所示核苷酸序列的sgrna。在另一優選例中,將釀酒酵母的染色體chrix和chrii進行融合,其中,所述著絲粒敲除組件用於敲除chrix著絲粒,並且,所述的著絲粒兩側同源序列分別為左同源臂chrix:355547-355606、右同源臂chrix:355840-355891,所述正向重複序列為chrix:355892-356106;所述染色體融合組件中,以chrix:435934-436360和chrii:8680-9089分別作為兩條染色體的近端粒區的同源序列,所述正向重複序列為chrii:9090-9359;所述sgrna靶向切割組件含有seqidno:6、seqidno:7、seqidno:8(對應於target4~6)所示核苷酸序列的sgrna。在另一優選例中,採用所述方法對酵母的染色體a和染色體b進行融合的同時,還包括:在同一體系中,採用所述方法將染色體a和染色體b的融合染色體與其它一個或多個染色體(如染色體c)進行融合。在另一優選例中,在同一體系中,將釀酒酵母的染色體chrvi,chri和chrii進行融合,其中,一個著絲粒敲除組件用於敲除chrvi著絲粒,並且,所述的著絲粒兩側同源序列分別為左同源臂chrvi:148410-148459、右同源臂chrvi:148725-148774,所述正向重複序列為chrvi:148775-149002;另一著絲粒敲除組件用於敲除chrii著絲粒,並且,所述的著絲粒兩側同源序列分別為左同源臂chrii:237974-238026、右同源臂chrii:238390-238439,所述正向重複序列為chrii:238440-238652;一個染色體融合組件針對chrvi和chri的融合,其中,兩條染色體的近端粒區的同源序列分別為chrvi:269212-269615和chri:2893-3294,所述正向重複序列為chri:3295-3519;另一染色體融合組件針對chri和chrii的融合,其中,兩條染色體的近端粒區的同源序列分別為chri:202775-203182和chrii:8680-9089,所述正向重複序列為chrii:9090-9359;所述sgrna靶向切割組件含有seqidno:3、seqidno:4、seqidno:5、seqidno:9、seqidno:10、seqidno:11所示核苷酸序列的sgrna。在另一優選例中,採用所述方法對酵母的染色體a和染色體b進行融合的同時,還包括:在同一體系中,採用同樣的方法對其它的一組或多組染色體(如染色體a』和b』)進行融合。在另一優選例中,在同一體系中,將釀酒酵母的染色體chrvi和chri融合,同時將chrix和chrii融合。在另一優選例中,與其它染色體進行融合時,融合的染色體僅保留一個著絲粒。在另一優選例中,表達cas9蛋白的酵母通過在酵母細胞中引入表達cas9的表達載體建立。在本發明的另一方面,提供所述方法的應用,應用於:(a)將酵母中兩條染色體融合為一條融合染色體;(b)將酵母中兩條以上的染色體融合為一條融合染色體;較佳地,該種融合在一個轉化子系統中進行;(c)將酵母中多條染色體進行兩兩融合;或(d)將酵母中多條染色體進行隨機組合的融合。在本發明的另一方面,提供一種用於對酵母的染色體進行融合的cas9表達組件,其是表達載體,其在酵母中為單拷貝複製、在大腸桿菌中為高拷貝複製,其中含有:酵母強啟動子以及由該啟動子驅動表達的cas9基因;較佳地,所述的強啟動子為tef1啟動子;來源於cen6ars4的載體複製區;篩選標記leu2;複製起始位點pbr322origin。在另一優選例中,所述的cas9表達組件的骨架載體具有seqidno:1所示的核酸序列。在本發明的另一方面,提供一種用於對酵母的染色體進行融合的sgrna靶向切割組件,其是能在酵母中組成型表達sgrna的表達載體,其中含有:酵母強啟動子(一個或多個)以及由該啟動子驅動表達的sgrna(一個或多個);較佳地,所述的強啟動子為snr52;載體複製區2micron2origi;酵母篩選標記his3;大腸甘菊篩選標記氨苄青黴素抗性基因;複製起始位點為pbr322origin。本發明的其它方面由於本文的公開內容,對本領域的技術人員而言是顯而易見的。附圖說明圖1、crispr/cas9介導的染色體融合方法原理圖。圖2、cas9表達質粒pleucas9示意圖。圖3、sgrna表達質粒psgrna示意圖。圖4、chrvi,chri融合脈衝電泳驗證。脈衝條件:1%的瓊脂糖凝膠,0.5*tbe緩衝液,12.5℃,轉換角度120°,電壓6v/cm,先60s轉換時間電泳22小時,再90s轉換時間電泳12小時。圖5、chrix,chrii融合脈衝電泳驗證。脈衝條件:1%的瓊脂糖凝膠,0.5*tbe緩衝液,12.5℃,轉換角度120°,電壓6v/cm,先60s轉換時間電泳22小時,再90s轉換時間電泳12小時。圖6、chrvi,chri,chrii融合脈衝電泳驗證。脈衝條件:1%的瓊脂糖凝膠,0.5*tbe緩衝液,12.5℃,轉換角度120°,電壓6v/cm,先60s轉換時間電泳22小時,再90s轉換時間電泳12小時。圖7、chrvi,chri和chrix,chrii同時融合脈衝電泳驗證。脈衝條件:1%的瓊脂糖凝膠,0.5*tbe緩衝液,12.5℃,轉換角度120°,電壓6v/cm,先60s轉換時間電泳22小時,再90s轉換時間電泳12小時。具體實施方式本發明人經過深入的研究,開發了一種利用crispr/cas9系統和酵母體內同源重組系統,進行酵母染色體融合的方法。本發明的方法可實現快速、高效的染色體融合。術語本發明中,所述的「核酸」可以指dna或rna。本文所用的術語「核酸序列」可以指dna或rna的序列。核酸序列可為環形。dna包括cdna、基因組dna或人工合成的dna。dna可以是單鏈的或是雙鏈的。dna可以是編碼鏈或非編碼鏈。例如,編碼區序列可以是seqidno:1所示的編碼序列的簡併變異體。此處所用的「簡併變異體」指與參比核酸編碼相同胺基酸序列、但核酸序列不同於參比核酸的核酸變異體。本發明中,所述的「載體」和「表達載體」可互換使用,指本領域熟知的細菌質粒、噬菌體、酵母質粒、植物細胞病毒、哺乳動物細胞病毒如腺病毒、逆轉錄病毒或其他載體。這些載體能夠在宿主體內複製和穩定。這些載體的一個重要特徵是通常含有複製起點、啟動子、標記基因和翻譯控制元件。本發明中,所述的「著絲粒敲除組件」是指含有一系列元件的核酸,其用於在進行染色體融合時,敲除多餘的著絲粒,使得融合後的染色體僅含有一個著絲粒。所述的「著絲粒敲除組件」含有待敲除著絲粒的兩側同源序列(同源臂)和篩選標記。所述的「著絲粒敲除組件」還含有必要的「調控元件」,從而可以被引入細胞內以及在細胞內複製、表達。本發明中,所述的「染色體融合組件」是指含有一系列元件的核酸,其用於待融合的染色體在合適的部位(通常位於端粒區)被識別和切割後,使得待融合的染色體發生融合;所述的「染色體融合組件」含有待融合的染色體的近端粒區的同源序列和篩選標記。所述的「染色體融合組件」還含有必要的「調控元件」,從而可以被引入細胞內以及在細胞內複製、表達。本發明中,所述的「sgrna靶向切割組件」是指用於在細胞內生成靶向特定位點的sgrna的核酸,其產生的sgrna能在著絲粒兩個同源序列間和近端粒區的兩個同源序列處進行切割,配合「著絲粒敲除組件」、「染色體融合組件」實現染色體的融合。所述的「sgrna靶向切割組件」還含有必要的「調控元件」,從而可以被引入細胞內以及在細胞內複製、表達。本發明中,所述的「調控元件」可包括啟動子、增強子、響應元件、信號肽、內部核糖體進入序列、聚腺苷酸信號、終止子、或調控核酸表達(如轉錄或翻譯)的可誘導元件。本文所用術語「基因組」指包含在一個生物體的dna(部分病毒是rna)中的全部遺傳信息。基因組包括基因和非編碼dna。一般地,一個生物體的基因組是指一套染色體中的完整的dna序列。例如,生物個體體細胞中的二倍體由兩套染色體組成,其中一套dna序列就是一個基因組。基因組一詞可特指核dna(例如,核基因組),也可用於包含自身dna序列的細胞器基因組,如粒線體基因組或葉綠體基因組。本文所述「敲除」或「刪除」指將目標基因從基因組中刪除的技術。本文所用術語「骨架載體」或「骨架質粒」指載體中除目標片段以外的部分。如本文所用,術語「包含」、「包括」和其等同形式包括「含有」以及「由……組成」的含義,例如「包含」x的組合物可僅由x組成或可含有其它物質,例如x+y。融合原理本發明提供了一種利用crispr/cas9系統在酵母中高效融合染色體的方法,該方法的原理及流程如圖1。通過酵母原生質體轉化技術,將含有著絲粒兩側同源序列和篩選標記的組件、參與融合的染色體兩個近端粒區的同源序列和篩選標記的組件以及能在著絲粒兩個同源序列間和近端粒區的兩個同源序列處發生切割的sgrna質粒一次轉化到酵母細胞內。染色體在著絲粒和端粒附近被切割後,含有染色體上同源序列的著絲粒敲除組件和染色體融合組件通過酵母體內的同源重組整合到染色體上,通過sgrna載體,cas9載體和著絲粒組件、染色體融合組件上的標記篩選出需要的轉化子。以往的染色體操作,往往受限於近端粒區有大段重複序列的染色體,而本發明人的方法可以實現任意染色體的融合;可以融合3條染色體為一條、4條染色體為兩條;可以實現無痕融合,即染色體上不殘留篩選標記;本發明的方法快速,簡便,可應用於其他具有高效重組系統的真核生物,實施染色體工程改造。融合方法本發明提供了一種對酵母的染色體a和染色體b進行融合的方法,所述方法包括:將著絲粒敲除組件、染色體融合組件、sgrna靶向切割組件、cas9表達組件引入到酵母中,從而獲得染色體a和染色體b發生融合的酵母。所述著絲粒敲除組件用於敲除兩條染色體之一的著絲粒,其含有該著絲粒兩側同源序列和篩選標記。當靶向性的sgrna在著絲粒的一側進行切割後,所述的著絲粒敲除組件通過同源交換,整合到染色體上。為了篩選發生交換的細胞,通常需要在著絲粒敲除組件中加上篩選標記。根據需要,所述的篩選標記可以是抗性基因,或者也可以是一段易於被識別的核酸片段。作為本發明的優選方式,在著絲粒敲除組件中,設置一段正向重複序列,其設置於該組件中所述同源序列與篩選標記之間;其序列為染色體上緊挨該著絲粒一側同源序列的序列,設置該正向重複序列,有利於將來在該區段進行進一步重組操作以消除篩選標記,獲得無痕融合的染色體。所述染色體融合組件在兩條染色體端粒附近發生雙交換,刪除端粒同時介導染色體融合,含有染色體a近端粒區的同源序列、染色體b近端粒區的同源序列和篩選標記。當靶向性的sgrna在要刪除的兩個端粒附近進行切割後,所述的染色體融合組件通過同源交換,整合到染色體,將兩條染色體連接在一起。為了篩選發生染色體融合的細胞,通常需要在著絲粒敲除組件中加上篩選標記。作為本發明的優選方式,所述染色體融合組件中,設置一段正向重複序列,設置於該組件中所述同源序列與篩選標記之間;其序列為緊挨著其中一條染色體近端粒區同源序列的序列。所述sgrna靶向切割組件含有能在著絲粒兩個同源序列間和近端粒區的兩個同源序列處進行切割的sgrna。可將多個sgrna串聯在一個質粒中進行表達,以簡化操作。作為本發明的優選方式,所述sgrna靶向切割組件是能在酵母中組成型表達sgrna的表達載體;所述sgrna靶向切割組件是能在酵母中組成型表達sgrna的表達載體,其在酵母中為單拷貝複製、在大腸桿菌中為高拷貝複製。較佳地,其以酵母強啟動子驅動sgrna的表達;更佳地,所述的強啟動子為snr52。更優選地,所述的表達載體的載體複製區為2micron2origi(來源於2μm質粒),以his3作為酵母表達的篩選標記;以氨苄青黴素抗性基因作為大腸桿菌表達的篩選標記,以pbr322origin(來源於pbr322)載體複製起始位點。作為本發明的優選方式,所述的cas9表達組件是表達載體,其中以酵母強啟動子驅動cas9基因的表達。例如,所述的強啟動子可以為tef1啟動子。所述的cas9表達組件在酵母中為單拷貝複製、在大腸桿菌中為高拷貝複製;更優選地,所述的表達載體的載體複製區來源於cen6ars4,以leu2作為篩選標記,以pbr322origin(來源於pbr322)作為複製起始位點。進一步優選地,所述的cas9表達組件的骨架載體具有seqidno:1所示的核酸序列。所述的cas9表達組件能夠高效地驅動cas9基因的表達,實現有效的基因編輯。利用本發明,可以直接將酵母的兩條染色體融合為一條。例如,本發明的實施例中,將釀酒酵母染色體chrvi,270kbp和chri,230kbp融合得到chrvi-i,503kbp;將染色體chrix,440kbp和chrii,810kbp融合得到chrix-ii,1247kbp)。利用本發明,可以在一次操作體系中將酵母的三條染色體融合為一條。例如,本發明的實施例中,將釀酒酵母的染色體chrvi,270kbp、chri,230kbp和chrii,810kbp融合得到chrvi-i-ii,1283kbp。利用本發明,可以在一次操作體系中將酵母的兩組兩條染色體進行融合。例如,本發明的實施例中,將釀酒酵母的染色體chrvi,270kbp和chri,230kbp融合得到chrvi-i,503kbp;同時將chrix,440kbp和chrii,810kbp融合得到chrix-ii,1247kbp。與常規的方法相比較,本發明可以融合任意兩條染色體,包括在近端粒區有大段其他染色體高度相似序列的染色體。在融合兩條染色體時,省去了先引入失活著絲粒組件再轉入染色體融合組件同時誘導著絲粒失活等繁瑣操作,比傳統方法更快速、高效。並且,本發明的方法還可以一次融合三條染色體、一次融合兩組兩條染色體。與傳統方法相比,不僅可以自由選擇融合的染色體,操作步驟更加簡便、大大縮短了實驗時間,還可一次融合更多數目的染色體,極為顯著地提高了染色體融合的效率。利用本方法可以逐步將釀酒酵母的16條染色體人工融合成1條,構建基因組更簡約的異源表達宿主和dna組裝「工廠」。下面結合具體實施例,進一步闡述本發明。應理解,這些實施例僅用於說明本發明而不用於限制本發明的範圍。下列實施例中未註明具體條件的實驗方法,通常按照常規條件如j.薩姆布魯克等編著,分子克隆實驗指南,第三版,科學出版社,2002中所述的條件,或按照製造廠商所建議的條件。sgrna識別序列實施例中所用的sgrna識別序列如表1所示。表1、sgrna識別序列名稱20bp序列pam位點target1識別位點(s1)tcgaacgtgatcctaacgag(seqidno:3)tggtarget2識別位點(s2)acacacgtttacgatgatat(seqidno:4)tggtarget3識別位點(s3)acggctaactgaacctaagt(seqidno:5)aggtarget4識別位點(s4)aaggagacaaattcagcgag(seqidno:6)tggtarget5識別位點(s5)tttcacgaatacgagataca(seqidno:7)gggtarget6識別位點(s6)agcgtagttaacccctctgc(seqidno:8)aggtarget7識別位點(s7)ctggtaaagagctctgcagc(seqidno:9)aggtarget8識別位點(s8)agcgtagttaacccctctgc(seqidno:10)aggtarget9識別位點(s9)taggatacaagtgtctgtgc(seqidno:11)aggcas9表達載體的構建cas9表達載體的質粒圖譜見圖2。cas9表達載體的序列見seqidno:1。構建在大腸桿菌中為高拷貝複製(複製起始位點來源於pbr322,篩選標記為氨苄青黴素(ampicillin)抗性基因)、在酵母中單拷貝複製(載體複製區來源於cen6ars4,篩選標記leu2)且能組成型表達cas9的質粒pleucas9。pcr擴增leu2標記(以釀酒酵母s.cerevisiaes288c基因組為模板),通過正反向引物的5』端帶上質粒pmetcas9上篩選標記met14(zhou,j.,etal.,nucleicacidsres,2016.44(14):p.e124)兩側50bp的同源序列,pcr產物純化後和pmetcas9一起轉入酵母,在亮氨酸單缺培養基上篩選,經pcr及測序驗證正確後轉化大腸桿菌dh10b,抽取質粒備用。sgrna表達載體構建sgrna表達載體的質粒圖譜見圖3。構建在大腸桿菌中為高拷貝複製(複製起始位點來源於pbr322,篩選標記為氨苄青黴素抗性基因)、在酵母中多拷貝複製(載體複製區來源於2μm,篩選標記his3)且能組成型表達sgrna的質粒phisgrna。sgrna包括snr52啟動子、handlecas9序列、20bp切割位點。chrvi和chri融合獲得chrvi-i需要target1,2,3三個切割位點,分別位於chrvi的著絲粒、右端粒和chri的左端粒附近。chrix和chrii融合獲得chrix-ii需要target4,5,6三個切割位點,分別位於chrix的著絲粒、右端粒和chrii的左端粒附近。sgrna載體的構建首先以p426-snr52p-grna.y-sup4t(agren,r.等,jindmicrobiolbiotechnol,2013.40(7):p.735-47)為模板,pcr切割位點grna.y以外的上下遊序列。通過引物帶上20bp切割位點、上下遊40bp重複序列和酶切位點,經融合pcr將grna的兩部分融合到一起獲得400bp完整的grna。target1,4的pcr產物用ecori、bamhi酶切,target2,5的pcr產物用bamhi、ncoi酶切,target3,6的pcr產物用ncoi、noti酶切,酶切好的target1,2,3和target4,5,6連到ecori、noti酶切的載體phisgrna((由質粒p426-snr52p-grna.y-sup4t質粒改造,將ura3篩選標記改為his3篩選標記)上。轉化大腸桿菌後抽質粒測序備用。chrvi,chri和chrii融合獲得chrvi-i-ii需要target1,2,3,7,8,9六個切割位點,分別位於chrvi著絲粒、右端粒,chri左端粒、右端粒和chrii著絲粒、左端粒附近。一次融合chrvi,chri和chrix,chrii兩組兩條染色體獲得chrvi-i、chrix-ii,需要target1,2,3,4,5,6六個切割位點。sgrna載體的構建首先以p426-snr52p-grna.y-sup4t為模板,pcr切割位點grna.y以外的上下遊序列。分別通過引物帶上20bp切割位點、上下遊40bp重複序列和golden-gate克隆所需的四個鹼基,通過融合pcr獲得的400bpgrna片段。將400bpgrna片段克隆到puc57(篩選標記為氨苄青黴素(ampicillin)抗性基因)上,測序正確後target1,2,3grna通過bsai酶切和t4連接酶連接作用(golden-gate)克隆到線性化載體phx31(篩選標記為阿普拉黴素(apramycin)抗性基因,含酶切位點ecori、bamhi和golden-gate克隆所需的四個鹼基)上。target4,5,6和target7,8,9grna通過bsai酶切和t4連接酶連接作用(golden-gate)克隆到線性化載體phx31(篩選標記為阿普拉黴素(apramycin)抗性基因,含酶切位點bamhi、noti和golden-gate克隆所需的四個鹼基)上。之後用ecori、bamhi酶切獲得target123,bamhi、noti酶切獲得target456,再將酶切片段target123和target456連接到ecori、noti酶切的phisgrna上獲得一次融合chrvi,chri和chrix,chrii兩組兩條染色體需要的sgrna;ecori、bamhi酶切target123和bamhi、noti酶切的target789連接到ecori、noti酶切的phisgrna上獲得一次融合chrvi,chri和chrii三條染色體需要的sgrna載體。質粒pgrna1-3含有啟動子snr52使sgrnatarget1、target2和target3可在酵母中組成型表達(在一次融合三條染色體和兩組兩條染色體實驗中質粒psgrna含有啟動子snr52使6個target位點可在酵母中組成型表達)。該質粒含酵母複製區(來源於2μorigin)和篩選標記(his3),在酵母中為多拷貝複製;同時含大腸桿菌複製區(來源於pbr322)和篩選標記氨苄青黴素(ampicillin),在大腸桿菌中為高拷貝複製。序列見segidno:2。質粒pgrna4-6含有啟動子snr52使sgrnatarget4、target5和target6可在酵母中組成型表達。著絲粒敲除組件的構建以by4742基因組為模板選取著絲粒左右兩側50bp序列作為敲除組件的左右同源臂,著絲粒一側200bp序列作為打靶組件上的正向重複序列(以便日後該序列處發生重組消除篩選標記,獲得無痕融合的染色體)。以s288c基因組為模板pcrura3基因作為篩選標記。將50bp同源臂序列帶在引物上,分別pcr正向重複序列和篩選標記,經融合pcr獲得著絲粒敲除組件。染色體融合組件的構建以by4742基因組為模板pcr一條染色體右端粒左側、另一條染色體左端粒右側的400bp序列作為染色體融合組件的同源臂,pcr一個端粒附近200bp序列作為融合組件上的正向重複序列(以便日後在該序列處發生重組消除篩選標記,獲得無痕融合的染色體)。以s288c基因組為模板pcrlys2基因作為篩選標記。四個片段間通過引物帶上40bp同源序列,經融合pcr獲得染色體融合組件。原生質體轉化將1μg的pgrna載體、1μg著絲粒敲除組件、1μg染色體融合組件用原生質體轉化的方法轉入酵母,在pgrna和cas9的共同作用下,在著絲粒、左/右端粒處發生切割。通過酵母體內高效的同源重組系統,含有酵母染色體上同源序列的著絲粒敲除組件和染色體融合組件雙交換到染色體上。通過共同篩選pcas9、pgrna、著絲粒敲除組件和染色體融合組件所帶的營養缺陷標記,得到需要的轉化重組子。陽性克隆經pcr、測序以及脈衝場凝膠電泳驗證。酵母原生質體製備和轉化的具體方法參考kouprina,n等,natprotoc,2008.3(3):p.371-7,在此基礎上本發明人做了改進,從而有效提高轉化效率,其步驟為:1.by4742/pleucas9在sc-leu平板上劃線,30℃培養2天;2.接單克隆到4mlsc-leu液體培養基中,30℃培養過夜;3.將過夜培養的菌液轉接到含50mlsc-leu液體培養基搖瓶中,調起始odλ=600nm=0.1;4.240rpm,30℃培養至終odλ=600nm=1.0;5.將菌液轉移到50ml無菌離心管中,5℃,1000g離心5分鐘收菌;6.棄上清,加30ml無菌水重懸菌體,5℃,3000g離心5分鐘收菌;7.棄上清,加20ml1m山梨醇溶液重懸菌體,5℃,3000g離心5分鐘收菌;8.棄上清,加20mlspe溶液重懸菌體,再加入20μl裂解酶融合和40μl巰基乙醇,混勻後30℃水浴鍋中孵育20分鐘;9.5℃,570g離心5分鐘收集原生質體;10.棄上清,用50ml1m山梨醇溶液輕柔混勻、重懸原生質體,570g離心10分鐘收集原生質體;11.棄上清,加50ml1m山梨醇溶液重複洗滌一次;12.加1.2mlstc溶液輕柔重懸原生質體;13.取200μl原生質體重懸液與1μgtarget123-phisgrna、1μg著絲粒敲除組件、1μg染色體融合組件(事先混勻,總體積約20μl)混勻;14.再加入800μlpeg8000,,輕柔倒轉混勻,室溫放置10分鐘;15.5℃,500g離心5分鐘收集原生質體;16.棄上清,加800μlsos重懸,30℃水浴鍋中孵育1小時;17.將原生質體轉移到sc-leu-his-ura-lys選擇培養基中(50℃平衡好),輕柔顛倒混勻後快速倒到sc-leu-his-ura-lys平板上;18.30℃培養3-4天直至所有的轉化子清晰可見;挑取4個轉化子通過菌落pcr進行初步驗證。菌落pcr具體步驟:1)引物設計,在著絲粒敲除組件同源臂外側設計驗證引物,如果著絲粒敲除成功,驗證引物pcr出2kbp左右條帶,如果不成功,驗證引物pcr出1kbp左右條帶;在染色體發生融合的位置設計外部驗證引物和內部驗證引物,在篩選標記lys2內部設計引物,如果染色體融合成功,由外部驗證引物和lys2內部引物pcr出1.5/1.0kbp條帶,如果不成功,由染色體上的外部驗證引物和內部驗證引物pcr出0.7kbp條帶。2)pcr,挑取酵母轉化子懸於15μl無菌水中,混勻後取1ul為模板,用kod-fx酶進行pcr驗證,pcr擴增條件為:94℃預變性4min;98℃變性10s,50℃退火30s,68℃延伸3min,35個循環。進行了兩次試驗,其中chrvi和chri融合轉化子數分別是150,634,菌落pcr驗證陽性率分別是4/4,4/4。其中chrix和chrii融合轉化子數分別是672,832,菌落pcr驗證陽性率分別是4/4,4/4。挑選兩個pcr驗證正確並測序正確的酵母準備製作膠塊,進行脈衝場凝膠電泳檢測。基本步驟為:1.從平板上挑取單克隆接ypad培養基,30℃,240rpm過夜培養;2.把試管內過夜培養的酵母轉接到50mlypad培養基中,起始odλ=600nm=0.1;3.30℃,240rpm培養至odλ=600nm=1.0時20℃,5,000rpm離心5分鐘收菌;4.50mlddh2o重懸酵母,20℃,5,000rpm離心5分鐘收菌;5.10mlph8.0,10mmedta重懸酵母,20℃,5,000rpm離心5分鐘收菌;6.750μlph7.2,10mm的tris.hcl重懸,轉到1.5mlep管中,20℃,5,000rpm離心5分鐘收菌;7.150μlph7.2,10mm的tris.hcl重懸,放到50℃水浴鍋中平衡;8.加入150μlzymolyse-20t溶液(20mg/mlzymolyse-20t,50%甘油,2.5%葡萄糖,50mmph8.0tris.hcl)和225μl2%的te25s溶解的低熔點瓊脂糖(事先配好,在50℃水浴鍋中平衡),混勻後倒入模具,冰箱4℃冷卻;9.30分鐘後取出膠塊,加入5mllyticasebuffer(ph7.5,10mm的tris.hcl和ph8.0,50mmedta)和500μlzymolyse-20t,37℃孵育3小時;10.25mlddh2o洗一次膠塊,然後用washbuffer(ph7.5,20mm的tris.hcl和ph8.0,50mmedta)洗滌一次;11.每1ml膠塊加入5ml蛋白酶反應液(ph8.0,100mmedta,0.2%sodiumdeoxycholate,1%sodiumlaurylsarcosine,1mg/ml的蛋白酶k)50℃消化36小時;12.50mlwashbuffer洗滌膠塊4次,於室溫下輕柔震蕩,每次30-60分鐘。13.使用bio-radchefdrii儀器,用1%的瓊脂糖凝膠在0.5*tbe緩衝液中12.5℃條件下進行分離。轉換角度設置為120°,電壓設置為6v/cm,先60s轉換時間電泳22小時,再90s轉換時間電泳12小時。實施例1、兩條染色體融合以釀酒酵母by4742為出發菌株,選取chrvi(270kbp)和chri(230kbp)、chrix(440kbp)和chrii(813kbp)進行融合(表2)。表2、兩條染色體融合信息將cas9表達質粒pleucas9轉入by4742酵母細胞中獲得菌株by4742/pleucas9。質粒pleucas9含有啟動子tef1使cas9可在酵母中組成型表達。該質粒含酵母複製區(cen6ars4)和篩選標記(leu2),在酵母中為單拷貝複製;同時含大腸桿菌複製區(來源於pbr322)和篩選標記氨苄青黴素(ampicillin),在大腸桿菌中為高拷貝複製。chrvi,chri融合時,敲除chrvi著絲粒,選取chrvi上148410-148459bp、148725-148774bp、148775-149002bp作為著絲粒敲除的左同源臂、右同源臂、正向重複序列(dr1),269212-269615bp作為染色體融合組件的左同源臂。chri上2893-3294bp、3295-3519bp作為染色體融合組件的右同源臂和正向重複序列(dr2)。將pgrna1-3、chrvi著絲粒敲除組件(篩選標記ura3)和chrvi-i融合組件(篩選標記lys2)轉化by4742/pleucas9原生質體。chrix,chrii融合時,敲除chrix著絲粒,選取chrix上355547-355606bp、355840-355891bp、355892-356106bp作為著絲粒敲除的左同源臂、右同源臂、正向重複序列,435934-436360bp作為染色體融合組件的左同源臂。chrii上8680-9089bp、9090-9359bp作為染色體融合組件的右同源臂和正向重複序列。將pgrna4-6、chrix著絲粒敲除組件(篩選標記ura3)和chrix-ii融合組件(篩選標記lys2)轉化by4742/pleucas9原生質體。脈衝場凝膠電泳結果顯示,chrvi(270kbp)和chri(230kbp)融合為一條503kbp的染色體(圖4),即chrvi-i。脈衝條件:1%的瓊脂糖凝膠,0.5*tbe緩衝液,12.5℃,轉換角度120°,電壓6v/cm,先60s轉換時間電泳22小時,再90s轉換時間電泳12小時。脈衝電泳圖顯示chrix(440kbp)和chrii(813kbp)條帶消失,多出一條1247kbp左右條帶,即chrix-ii。因此,chrix(440kbp)和chrii(813kbp)融合為一條1247kbp的染色體(圖5)。實施例2、三條染色體融合本實施例中,以釀酒酵母by4742為出發菌株,選取chrvi(270kbp)和chri(230kbp)和chrii(813kbp)進行融合,見表3。表3、三條染色體融合信息chrvi(270kbp)、chri(230kbp)和chrii(813kbp)融合時,敲除chrvi和chrii的著絲粒。選取chrvi上148410-148459bp、148725-148774bp、148775-149002bp作為著絲粒敲除的左同源臂、右同源臂、正向重複序列;269212-269615bp作為chrvi-i融合組件的左同源臂。chri上2893-3294bp、3295-3519bp作為chrvi-i融合組件的右同源臂和正向重複序列;202775-203182bp作為chri-ii融合組件左同源臂。chrii上8680-9089bp、9090-9359bp作為chri-ii融合組件的右同源臂和正向重複序列。選取chrii上237974-238026bp、238390-238439bp、238440-238652bp作為著絲粒敲除的左同源臂、右同源臂、正向重複序列。將pgrna123789(見表1)、chrvi著絲粒敲除組件(ura3)、chrvi-i融合組件(lys2)、chri-ii融合組件(natmx6,nourseothricin)、chrii著絲粒敲除組件(ura3)用實施例1中所述原生質體轉化的方法轉移到by4742/pleucas9中。挑取4個轉化子通過菌落pcr進行初步驗證。菌落pcr具體步驟:1)引物設計,在chrvi、chrii著絲粒同源臂外側設計驗證引物,如果著絲粒敲除成功,驗證引物pcr出2kbp左右條帶,如果不成功,驗證引物pcr出1kbp左右條帶;在chrvi、chri發生融合的位置設計外部驗證引物和內部驗證引物和lys2上的驗證引物,如果染色體融合成功,則由外部驗證引物和lys2上的驗證引物pcr出1.5/1.0kbp條帶,如果融合不成功,則由染色體上的外部驗證引物和內部驗證引物pcr出0.7kbp條帶。在chri、chrii發生融合的位置設計外部驗證引物和內部驗證引物,如果融合成功,則由兩條外部驗證引物pcr出2.5kbp條帶,如果失敗,則由一條外部驗證引物和內部驗證引物pcr出1.0kbp條帶。進行了兩次試驗,融合轉化子數分別是12,7,菌落pcr驗證陽性率分別是1/4,1/4。按實施例1中所述菌落pcr方法進行初步驗證,挑一個正確的克隆按實例一中所述進行脈衝電泳驗證。脈衝電泳圖顯示chrvi(270kbp)、chri(230kbp)和chrii(813kbp)條帶消失,多出一條1283kbp左右條帶,即chrvi-i-ii(圖6)。脈衝場凝膠電泳結果說明,chrvi(270kbp)、chri(230kbp)和chrii(813kbp)融合為一條1283kbp的染色體。實施例3、兩組兩條染色體融合本例中以釀酒酵母by4742為出發菌株,選取chrvi(270kbp)和chri(230kbp)、chrix(440kbp)和chrii(813kbp)同時融合(表4)。表4、兩組兩條染色體融合信息chrvi(270kbp)和chri(230kbp)、chrix(440kbp)和chrii(810kbp)同時融合時,將pgrna1-6、chrvi著絲粒敲除組件(ura3)、chrvi-i融合組件(ura3)、chrix著絲粒敲除組件(lys2)、chrix-ii融合組件(lys2)用實施例1中所述原生質體融合轉化的方法轉移到by4742/pleucas9中(同源臂的選擇參見實施例1)。按實施例1中所述菌落pcr方法進行初步驗證(驗證引物參見實施例1),進行了兩次試驗,融合轉化子數分別是10,24,菌落pcr驗證陽性率分別是3/4,3/4。挑一個正確的克隆按實施例1中所述進行脈衝電泳驗證。脈衝電泳圖顯示chrvi(270kbp)、chri(230kbp)和chrix(440kbp)、chrii(813kbp)條帶消失,多出一條500kbp左右條帶chrvi-i和一條1247kbp左右條帶chrix-ii。結果說明,chrvi(270kbp)、chri(230kbp)和chrix(440kbp)、chrii(813kbp)四條染色體同時兩兩融合為503kbp,1247kbp大小的染色體(見圖7)。通過酵母原生質體轉化技術,將著絲粒敲除組件,染色體融合組件以及pgrna質粒一次轉化到酵母細胞內。染色體在著絲粒和端粒附近被切割後,含有染色體上同源序列的著絲粒敲除組件和染色體融合組件通過酵母體內的同源重組整合到染色體上,通過sgrna載體,cas9載體和著絲粒組件、染色體融合組件上的標記篩選出需要的轉化子。因打靶組件上帶有可消除標記的正向重複序列,還可以誘導切割獲得無痕融合的染色體。在本發明提及的所有文獻都在本申請中引用作為參考,就如同每一篇文獻被單獨引用作為參考那樣。此外應理解,在閱讀了本發明的上述講授內容之後,本領域技術人員可以對本發明作各種改動或修改,這些等價形式同樣落於本申請所附權利要求書所限定的範圍。序列表中國科學院上海生命科學研究院一種高效的酵母菌染色體融合方法17501211siposequencelisting1.0110781dna人工序列(artificialsequence)misc_feature表達載體1gacgaaagggcctcgtgatacgcctatttttataggttaatgtcatgataataatggttt60cttaggacggatcgcttgcctgtaacttacacgcgcctcgtatcttttaatgatggaata120atttgggaatttactctgtgtttatttatttttatgttttgtatttggattttagaaagt180aaataaagaaggtagaagagttacggaatgaagaaaaaaaaataaacaaaggtttaaaaa240atttcaacaaaaagcgtactttacatatatatttattagacaagaaaagcagattaaata300gatatacattcgattaacgataagtaaaatgtaaaatcacaggattttcgtgtgtggtct360tctacacagacaagatgaaacaattcggcattaatacctgagagcaggaagagcaagata420aaaggtagtatttgttggcgatccccctagagtcttttacatcttcggaaaacaaaaact480attttttctttaatttctttttttactttctatttttaatttatatatttatattaaaaa540atttaaattataattatttttatagcacgtgatgaaaaggacccaggtggcacttttcgg600ggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccg660ctcatgagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagt720attcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgttttt780gctcacccagaaacgctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtg840ggttacatcgaactggatctcaacagcggtaagatccttgagagttttcgccccgaagaa900cgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtatt960gacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgag1020tactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagt1080gctgccataaccatgagtgataacactgcggccaacttacttctgacaacgatcggagga1140ccgaaggagctaaccgcttttttgcacaacatgggggatcatgtaactcgccttgatcgt1200tgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgta1260gcaatggcaacaacgttgcgcaaactattaactggcgaactacttactctagcttcccgg1320caacaattaatagactggatggaggcggataaagttgcaggaccacttctgcgctcggcc1380cttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggt1440atcattgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacg1500gggagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactg1560attaagcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaa1620cttcatttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaa1680atcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaagga1740tcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccg1800ctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaact1860ggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccac1920cacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtg1980gctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccg2040gataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcga2100acgacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttccc2160gaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacg2220agggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctc2280tgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgcc2340agcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttcttt2400cctgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgatacc2460gctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgc2520ccaatacgcaaaccgcctctccccgcgcgttggccgattcattaatgcagctggcacgac2580aggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatgtgagttacctcact2640cattaggcaccccaggctttacactttatgcttccggctcctatgttgtgtggaattgtg2700agcggataacaatttcacacaggaaacagctatgaccatgattacgccaagcgcgcaatt2760aaccctcactaaagggaacaaaagctggagctcgtgacagccctccgaaggaagactctc2820ctccgtgcgtcctcgtcttcaccggtcgcgttcctgaaacgcagatgtgcctcgcgccgc2880actgctccgaacaataaagattctacaatactagcttttatggttatgaagaggaaaaat2940tggcagtaacctggccccacaaaccttcaaatgaacgaatcaaattaacaaccataggat3000gataatgcgattagttttttagccttatttctggggtaattaatcagcgaagcgatgatt3060tttgatctattaacggatatataaatgcaaaaactgcataaccactttaactaatacttt3120caacattttcggtttgtattacttcttattcaaatgtaataaaagtatcaacaaaaaatt3180gttaatatacctctatactttaacgtcaaggagaaaaaaccccggattctagcttgcctt3240gtccccgccgggtcacccggccagcgacatggaggcccagaataccctccttgacagtct3300tgacgtgcgcagctcaggggcatgatgtgactgtcgcccgtacatttagcccatacatcc3360ccatgtataatcatttgcatccatacattttgatggccgcacggcgcgaagcaaaaatta3420cggctcctcgctgcagacctgcgagcagggaaacgctcccctcacagacgcgttgaattg3480tccccacgccgcgcccctgtagagaaatataaaaggttaggatttgccactgaggttctt3540ctttcatatacttccttttaaaatcttgctaggatacagttctcacatcacatccgaaca3600taaacaaccatggacaagaagtactccattgggctcgatatcggcacaaacagcgtcggc3660tgggccgtcattacggacgagtacaaggtgccgagcaaaaaattcaaagttctgggcaat3720accgatcgccacagcataaagaagaacctcattggcgccctcctgttcgactccggggag3780acggccgaagccacgcggctcaaaagaacagcacggcgcagatatacccgcagaaagaat3840cggatctgctacctgcaggagatctttagtaatgagatggctaaggtggatgactctttc3900ttccataggctggaggagtcctttttggtggaggaggataaaaagcacgagcgccaccca3960atctttggcaatatcgtggacgaggtggcgtaccatgaaaagtacccaaccatatatcat4020ctgaggaagaagcttgtagacagtactgataaggctgacttgcggttgatctatctcgcg4080ctggcgcatatgatcaaatttcggggacacttcctcatcgagggggacctgaacccagac4140aacagcgatgtcgacaaactctttatccaactggttcagacttacaatcagcttttcgaa4200gagaacccgatcaacgcatccggagttgacgccaaagcaatcctgagcgctaggctgtcc4260aaatcccggcggctcgaaaacctcatcgcacagctccctggggagaagaagaacggcctg4320tttggtaatcttatcgccctgtcactcgggctgacccccaactttaaatctaacttcgac4380ctggccgaagatgccaagcttcaactgagcaaagacacctacgatgatgatctcgacaat4440ctgctggcccagatcggcgaccagtacgcagacctttttttggcggcaaagaacctgtca4500gacgccattctgctgagtgatattctgcgagtgaacacggagatcaccaaagctccgctg4560agcgctagtatgatcaagcgctatgatgagcaccaccaagacttgactttgctgaaggcc4620cttgtcagacagcaactgcctgagaagtacaaggaaattttcttcgatcagtctaaaaat4680ggctacgccggatacattgacggcggagcaagccaggaggaattttacaaatttattaag4740cccatcttggaaaaaatggacggcaccgaggagctgctggtaaagcttaacagagaagat4800ctgttgcgcaaacagcgcactttcgacaatggaagcatcccccaccagattcacctgggc4860gaactgcacgctatcctcaggcggcaagaggatttctacccctttttgaaagataacagg4920gaaaagattgagaaaatcctcacatttcggataccctactatgtaggccccctcgcccgg4980ggaaattccagattcgcgtggatgactcgcaaatcagaagagaccatcactccctggaac5040ttcgaggaagtcgtggataagggggcctctgcccagtccttcatcgaaaggatgactaac5100tttgataaaaatctgcctaacgaaaaggtgcttcctaaacactctctgctgtacgagtac5160ttcacagtttataacgagctcaccaaggtcaaatacgtcacagaagggatgagaaagcca5220gcattcctgtctggagagcagaagaaagctatcgtggacctcctcttcaagacgaaccgg5280aaagttaccgtgaaacagctcaaagaagactatttcaaaaagattgaatgtttcgactct5340gttgaaatcagcggagtggaggatcgcttcaacgcatccctgggaacgtatcacgatctc5400ctgaaaatcattaaagacaaggacttcctggacaatgaggagaacgaggacattcttgag5460gacattgtcctcacccttacgttgtttgaagatagggagatgattgaagaacgcttgaaa5520acttacgctcatctcttcgacgacaaagtcatgaaacagctcaagaggcgccgatataca5580ggatgggggcggctgtcaagaaaactgatcaatgggatccgagacaagcagagtggaaag5640acaatcctggattttcttaagtccgatggatttgccaaccggaacttcatgcagttgatc5700catgatgactctctcacctttaaggaggacatccagaaagcacaagtttctggccagggg5760gacagtcttcacgagcacatcgctaatcttgcaggtagcccagctatcaaaaagggaata5820ctgcagaccgttaaggtcgtggatgaactcgtcaaagtaatgggaaggcataagcccgag5880aatatcgttatcgagatggcccgagagaaccaaactacccagaagggacagaagaacagt5940agggaaaggatgaagaggattgaagagggtataaaagaactggggtcccaaatccttaag6000gaacacccagttgaaaacacccagcttcagaatgagaagctctacctgtactacctgcag6060aacggcagggacatgtacgtggatcaggaactggacatcaatcggctctccgactacgac6120gtggatcatatcgtgccccagtcttttctcaaagatgattctattgataataaagtgttg6180acaagatccgataaaaatagagggaagagtgataacgtcccctcagaagaagttgtcaag6240aaaatgaaaaattattggcggcagctgctgaacgccaaactgatcacacaacggaagttc6300gataatctgactaaggctgaacgaggtggcctgtctgagttggataaagccggcttcatc6360aaaaggcagcttgttgagacacgccagatcaccaagcacgtggcccaaattctcgattca6420cgcatgaacaccaagtacgatgaaaatgacaaactgattcgagaggtgaaagttattact6480ctgaagtctaagctggtctcagatttcagaaaggactttcagttttataaggtgagagag6540atcaacaattaccaccatgcgcatgatgcctacctgaatgcagtggtaggcactgcactt6600atcaaaaaatatcccaagcttgaatctgaatttgtttacggagactataaagtgtacgat6660gttaggaaaatgatcgcaaagtctgagcaggaaataggcaaggccaccgctaagtacttc6720ttttacagcaatattatgaattttttcaagaccgagattacactggccaatggagagatt6780cggaagcgaccacttatcgaaacaaacggagaaacaggagaaatcgtgtgggacaagggt6840agggatttcgcgacagtccggaaggtcctgtccatgccgcaggtgaacatcgttaaaaag6900accgaagtacagaccggaggcttctccaaggaaagtatcctcccgaaaaggaacagcgac6960aagctgatcgcacgcaaaaaagattgggaccccaagaaatacggcggattcgattctcct7020acagtcgcttacagtgtactggttgtggccaaagtggagaaagggaagtctaaaaaactc7080aaaagcgtcaaggaactgctgggcatcacaatcatggagcgatcaagcttcgaaaaaaac7140cccatcgactttctcgaggcgaaaggatataaagaggtcaaaaaagacctcatcattaag7200cttcccaagtactctctctttgagcttgaaaacggccggaaacgaatgctcgctagtgcg7260ggcgagctgcagaaaggtaacgagctggcactgccctctaaatacgttaatttcttgtat7320ctggccagccactatgaaaagctcaaagggtctcccgaagataatgagcagaagcagctg7380ttcgtggaacaacacaaacactaccttgatgagatcatcgagcaaataagcgaattctcc7440aaaagagtgatcctcgccgacgctaacctcgataaggtgctttctgcttacaataagcac7500agggataagcccatcagggagcaggcagaaaacattatccacttgtttactctgaccaac7560ttgggcgcgcctgcagccttcaagtacttcgacaccaccatagacagaaagcggtacacc7620tctacaaaggaggtcctggacgccacactgattcatcagtcaattacggggctctatgaa7680acaagaatcgacctctctcagctcggtggagacagcagggctgaccccaagaagaagagg7740aaggtgtgattttggacctcgagtcattggacctcgagtcatgtaattagttatgtcacg7800cttacattcacgccctccccccacatccgctctaaccgaaaaggaaggagttagacaacc7860tgaagtctaggtccctatttatttttttatagttatgttagtattaagaacgttatttat7920atttcaaatttttcttttttttctgtacagacgcgtgtacgcatgtaacattatactgaa7980aaccttgcttgagaaggttttgggacgctcgaaggctttaatttgcggccggtacccaat8040tcgccctatagtgagtcgtattacgcgcgctcactggccgtcgttttacaacgtcgtgac8100tgggaaaaccctggcgttacccaacttaatcgccttgcagcacatccccctttcgccagc8160tggcgtaatagcgaagaggcccgcaccgatcgcccttcccaacagttgcgcagcctgaat8220ggcgaatggcgcgacgcgccctgtagcggcgcattaagcgcggcgggtgtggtggttacg8280cgcagcgtgaccgctacacttgccagcgccctagcgcccgctcctttcgctttcttccct8340tcctttctcgccacgttcgccggctttccccgtcaagctctaaatcgggggctcccttta8400gggttccgatttagtgctttacggcacctcgaccccaaaaaacttgattagggtgatggt8460tcacgtagtgggccatcgccctgatagacggtttttcgccctttgacgttggagtccacg8520ttctttaatagtggactcttgttccaaactggaacaacactcaaccctatctcggtctat8580tcttttgatttataagggattttgccgatttcggcctattggttaaaaaatgagctgatt8640taacaaaaatttaacgcgaattttaacaaaatattaacgtttacaatttcctgatgcggt8700attttctccttacgcatctgtgcggtatttcacaccgcatatcgacggtcgaggagaact8760tctagtatatccacatacctaatattattgccttattaaaaatggaatcccaacaattac8820atcaaaatccacattctcttcaaaatcaattgtcctgtacttccttgttcatgtgtgttc8880aaaaacgttatatttataggataattatactctatttctcaacaagtaattggttgtttg8940gccgagcggtctaaggcgcctgattcaagaaatatcttgaccgcaagattacgtttaaag9000gagcattaacaggtttactcataacaatcattttcaaatttccctatgcatgtttagagc9060aagcgcctttgtgagccctcccggttacgacgccttggcaatgtagcagataactctgca9120cttctagagctcgtgacagccctccgaaggaagactctcctccgtgcgtcctcgtcttca9180ccggtcgcgttcctgaaacgcagatgtgcctcgcgccgcactgctccgaacaataaagat9240tctacaatactagcttttatggttatgaagaggaaaaattggcagtaacctggccccaca9300aaccttcaaatgaacgaatcaaattaacaaccataggatgataatgcgattagtttttta9360gccttatttctggggtaattaatcagcgaagcgatgatttttgatctattaacggatata9420taaatgcaaaaactgcataaccactttaactaatactttcaacattttcggtttgtatta9480cttcttattcaaatgtaataaaagtatcaacaaaaaattgttaatatacctctatacttt9540aacgtcaaggagaaaaaaccccctgattactagcgaagctgcgttttagagctagaaata9600gcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtggtg9660ctttttttgttttttatgtcttcgagtcatgtaattagttatgtcacgcttacattcacg9720ccctccccccacatccgctctaaccgaaaaggaaggagttagacaacctgaagtctaggt9780ccctatttatttttttatagttatgttagtattaagaacgttatttatatttcaaatttt9840tcttttttttctgtacagacgcgtgtacgcatgtaacattatactgaaaaccttgcttga9900gaaggttttgggacgctcgaaggcttttctagaatcattccactacgacatttggctcat9960caccagctcgcgagaaatgtaaataagccaacaaccaagaatgcgtaacattaaagaata10020cagttgctttcatttcggcgtgatggtacggcacccacggtaccttacattattctcgaa10080aaatagctgcacgcttttccaggaataaaagaccgtgccactaatttcacgtgatcaata10140tatttacaagccacctcaaaaaatgtggcaatggagaagaggatgaacgactcaatatga10200ggaatatgttcatagggtagacgaaactatatacgcaatctacatacatttatcaagaag10260gagaaaaaggaggatagtaaaggaatacaggtaagcaaattgatactaatggctcaacgt10320gataaggaaaaagaattgcactttaacattaatattgacaaggaggagggcaccacacaa10380aaagttaggtgtaacagaaaatcatgaaactacgattcctaatttgatattggaggattt10440tctctaaaaaaaaaaaaatacaacaaataaaaaacactcaatgacctgaccatttgatgg10500agtttaagtcaataccttcttgaaccatttcccataatggtgaaagttccctcaagaatt10560ttactctgtcagaaacggccttacgacgtagtcgatatggtgcactctcagtacaatctg10620ctctgatgccgcatagttaagccagccccgacacccgccaacacccgctgacgcgccctg10680acgggcttgtctgctcccggcatccgcttacagacaagctgtgaccgtctccgggagctg10740catgtgtcagaggttttcaccgtcatcaccgaaacgcgcga1078127096dna人工序列(artificialsequence)misc_feature表達載體2gacgaaagggcctcgtgatacgcctatttttataggttaatgtcatgataataatggttt60cttagtatgatccaatatcaaaggaaatgatagcattgaaggatgagactaatccaattg120aggagtggcagcatatagaacagctaaagggtagtgctgaaggaagcatacgataccccg180catggaatgggataatatcacaggaggtactagactacctttcatcctacataaatagac240gcatataagtacgcatttaagcataaacacgcactatgccgttcttctcatgtatatata300tatacaggcaacacgcagatataggtgcgacgtgaacagtgagctgtatgtgcgcagctc360gcgttgcattttcggaagcgctcgttttcggaaacgctttgaagttcctattccgaagtt420cctattctctagaaagtataggaacttcagagcgcttttgaaaaccaaaagcgctctgaa480gacgcactttcaaaaaaccaaaaacgcaccggactgtaacgagctactaaaatattgcga540ataccgcttccacaaacattgctcaaaagtatctctttgctatatatctctgtgctatat600ccctatataacctacccatccacctttcgctccttgaacttgcatctaaactcgacctct660acattttttatgtttatctctagtattactctttagacaaaaaaattgtagtaagaacta720ttcatagagtgaatcgaaaacaatacgaaaatgtaaacatttcctatacgtagtatatag780agacaaaatagaagaaaccgttcataattttctgaccaatgaagaatcatcaacgctatc840actttctgttcacaaagtatgcgcaatccacatcggtatagaatataatcggggatgcct900ttatcttgaaaaaatgcacccgcagcttcgctagtaatcagtaaacgcgggaagtggagt960caggctttttttatggaagagaaaatagacaccaaagtagccttcttctaaccttaacgg1020acctacagtgcaaaaagttatcaagagactgcattatagagcgcacaaaggagaaaaaaa1080gtaatctaagatgctttgttagaaaaatagcgctctcgggatgcatttttgtagaacaaa1140aaagaagtatagattctttgttggtaaaatagcgctctcgcgttgcatttctgttctgta1200aaaatgcagctcagattctttgtttgaaaaattagcgctctcgcgttgcatttttgtttt1260acaaaaatgaagcacagattcttcgttggtaaaatagcgctttcgcgttgcatttctgtt1320ctgtaaaaatgcagctcagattctttgtttgaaaaattagcgctctcgcgttgcattttt1380gttctacaaaatgaagcacagatgcttcgttcaggtggcacttttcggggaaatgtgcgc1440ggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaa1500taaccctgataaatgcttcaataatattgaaaaaggaagagtatgagtattcaacatttc1560cgtgtcgcccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaa1620acgctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcgaa1680ctggatctcaacagcggtaagatccttgagagttttcgccccgaagaacgttttccaatg1740atgagcacttttaaagttctgctatgtggcgcggtattatcccgtattgacgccgggcaa1800gagcaactcggtcgccgcatacactattctcagaatgacttggttgagtactcaccagtc1860acagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataacc1920atgagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagcta1980accgcttttttgcacaacatgggggatcatgtaactcgccttgatcgttgggaaccggag2040ctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgtagcaatggcaaca2100acgttgcgcaaactattaactggcgaactacttactctagcttcccggcaacaattaata2160gactggatggaggcggataaagttgcaggaccacttctgcgctcggcccttccggctggc2220tggtttattgctgataaatctggagccggtgagcgtgggtctcgcggtatcattgcagca2280ctggggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtcaggca2340actatggatgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattgg2400taactgtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaa2460tttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgt2520gagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagat2580cctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtg2640gtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggcttcagcaga2700gcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaac2760tctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagt2820ggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcag2880cggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacacc2940gaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaag3000gcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttcca3060gggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgt3120cgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcc3180tttttacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcc3240cctgattctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagc3300cgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcccaatacgcaaa3360ccgcctctccccgcgcgttggccgattcattaatgcagctggcacgacaggtttcccgac3420tggaaagcgggcagtgagcgcaacgcaattaatgtgagttacctcactcattaggcaccc3480caggctttacactttatgcttccggctcctatgttgtgtggaattgtgagcggataacaa3540tttcacacaggaaacagctatgaccatgattacgccaagcgcgcaattaaccctcactaa3600agggaacaaaagctggagaattctctttgaaaagataatgtatgattatgctttcactca3660tatttatacagaaacttgatgttttctttcgagtatatacaaggtgattacatgtacgtt3720tgaagtacaactctagattttgtagtgccctcttgggctagcggtaaaggtgcgcatttt3780ttcacaccctacaatgttctgttcaaaagattttggtcaaacgctgtagaagtgaaagtt3840ggtgcgcatgtttcggcgttcgaaacttctccgcagtgaaagataaatgatctcgaacgt3900gatcctaacgaggttttagagctagaaatagcaagttaaaataaggctagtccgttatca3960acttgaaaaagtggcaccgagtcggtggtgctttttttgttttttatgtctggatcctct4020ttgaaaagataatgtatgattatgctttcactcatatttatacagaaacttgatgttttc4080tttcgagtatatacaaggtgattacatgtacgtttgaagtacaactctagattttgtagt4140gccctcttgggctagcggtaaaggtgcgcattttttcacaccctacaatgttctgttcaa4200aagattttggtcaaacgctgtagaagtgaaagttggtgcgcatgtttcggcgttcgaaac4260ttctccgcagtgaaagataaatgatcacacacgtttacgatgatatgttttagagctaga4320aatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggt4380ggtgctttttttgttttttatgtctccatggtctttgaaaagataatgtatgattatgct4440ttcactcatatttatacagaaacttgatgttttctttcgagtatatacaaggtgattaca4500tgtacgtttgaagtacaactctagattttgtagtgccctcttgggctagcggtaaaggtg4560cgcattttttcacaccctacaatgttctgttcaaaagattttggtcaaacgctgtagaag4620tgaaagttggtgcgcatgtttcggcgttcgaaacttctccgcagtgaaagataaatgatc4680acggctaactgaacctaagtgttttagagctagaaatagcaagttaaaataaggctagtc4740cgttatcaacttgaaaaagtggcaccgagtcggtggtgctttttttgttttttatgtctg4800cggccgcagtcatgtaattagttatgtcacgcttacattcacgccctccccccacatccg4860ctctaaccgaaaaggaaggagttagacaacctgaagtctaggtccctatttattttttta4920tagttatgttagtattaagaacgttatttatatttcaaatttttcttttttttctgtaca4980gacgcgtgtacgcatgtaacattatactgaaaaccttgcttgagaaggttttgggacgct5040cgaaggctttaatttgcggccggtacccaattcgccctatagtgagtcgtattacgcgcg5100ctcactggccgtcgttttacaacgtcgtgactgggaaaaccctggcgttacccaacttaa5160tcgccttgcagcacatccccctttcgccagctggcgtaatagcgaagaggcccgcaccga5220tcgcccttcccaacagttgcgcagcctgaatggcgaatggcgcgacgcgccctgtagcgg5280cgcattaagcgcggcgggtgtggtggttacgcgcagcgtgaccgctacacttgccagcgc5340cctagcgcccgctcctttcgctttcttcccttcctttctcgccacgttcgccggctttcc5400ccgtcaagctctaaatcgggggctccctttagggttccgatttagtgctttacggcacct5460cgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctgatagac5520ggtttttcgccctttgacgttggagtccacgttctttaatagtggactcttgttccaaac5580tggaacaacactcaaccctatctcggtctattcttttgatttataagggattttgccgat5640ttcggcctattggttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaa5700aatattaacgtttacaatttcctgatgcggtattttctccttacgcatctgtgcggtatt5760tcacaccgcatagggtctcgaggagcttggtgagcgctaggagtcactgccaggtatcgt5820ttgaacacggcattagtcagggaagtcataacacagtcctttcccgcaattttctttttc5880tattactcttggcctcctctagtacactctatatttttttatgcctcggtaatgattttc5940atttttttttttcccctagcggatgactctttttttttcttagcgattggcattatcaca6000taatgaattatacattatataaagtaatgtgatttcttcgaagaatatactaaaaaatga6060gcaggcaagataaacgaaggcaaagatgacagagcagaaagccctagtaaagcgtattac6120aaatgaaaccaagattcagattgcgatctctttaaagggtggtcccctagcgatagagca6180ctcgatcttcccagaaaaagaggcagaagcagtagcagaacaggccacacaatcgcaagt6240gattaacgtccacacaggtatagggtttctggaccatatgatacatgctctggccaagca6300ttccggctggtcgctaatcgttgagtgcattggtgacttacacatagacgaccatcacac6360cactgaagactgcgggattgctctcggtcaagcttttaaagaggccctactggcgcgtgg6420agtaaaaaggtttggatcaggatttgcgcctttggatgaggcactttccagagcggtggt6480agatctttcgaacaggccgtacgcagttgtcgaacttggtttgcaaagggagaaagtagg6540agatctctcttgcgagatgatcccgcattttcttgaaagctttgcagaggctagcagaat6600taccctccacgttgattgtctgcgaggcaagaatgatcatcaccgtagtgagagtgcgtt6660caaggctcttgcggttgccataagagaagccacctcgcccaatggtaccaacgatgttcc6720ctccaccaaaggtgttcttatgtagtgacaccgattatttaaagctgcagcatacgatat6780atatacatgtgtatatatgtatacctatgaatgtcagtaagtatgtatacgaacagtatg6840atactgaagatgacaaggtaatgcatcattctatacgtgtcattctgaagagctcattga6900aaagctgtggtatggtgcactctcagtacaatctgctctgatgccgcatagttaagccag6960ccccgacacccgccaacacccgctgacgcgccctgacgggcttgtctgctcccggcatcc7020gcttacagacaagctgtgaccgtctccgggagctgcatgtgtcagaggttttcaccgtca7080tcaccgaaacgcgcga7096320dna人工序列(artificialsequence)misc_featuresgrna識別序列3tcgaacgtgatcctaacgag20420dna人工序列(artificialsequence)misc_featuresgrna識別序列4acacacgtttacgatgatat20520dna人工序列(artificialsequence)misc_featuresgrna識別序列5acggctaactgaacctaagt20620dna人工序列(artificialsequence)misc_featuresgrna識別序列6aaggagacaaattcagcgag20720dna人工序列(artificialsequence)misc_feature引物misc_featuresgrna識別序列7tttcacgaatacgagataca20820dna人工序列(artificialsequence)misc_featuresgrna識別序列8agcgtagttaacccctctgc20920dna人工序列(artificialsequence)misc_featuresgrna識別序列9ctggtaaagagctctgcagc201020dna人工序列(artificialsequence)misc_featuresgrna識別序列10agcgtagttaacccctctgc201120dna人工序列(artificialsequence)misc_featuresgrna識別序列11taggatacaagtgtctgtgc20當前第1頁12
