一種非模式生物轉錄組基因序列結構分析的方法與流程
2023-05-29 08:58:01 2
本發明涉及基因分析生物信息學領域,具體是一種非模式生物轉錄組基因序列結構分析的方法。
背景技術:
基因是細胞內具有生物學功能的一段核酸序列,其翻譯的蛋白質直接參與了細胞的各種生理生化反應,是細胞賴以生存的基礎。近些年生命科學的研究的其中一個最主要的方向就是基因功能探索,所以很多生物基礎的研究的起點就是確定該物種的基因數目和類型。傳統的基因同源克隆為生物基因序列的獲得和研究提供了可靠的研究方法,但是基因克隆只能對單個基因序列逐一進行,很難對細胞內所有的基因序列進行高通量的測序。最新的高通量測序技術在轉錄組學上的應用,轉錄組測序為細胞內所有的基因序列的大規模測定提供了新的方法,但是這種大規模測序又為海量基因序列的準確分析提出了新的挑戰。如何確定轉錄組測序拼接得到的大量基因,特別是蛋白編碼基因的序列結構是後續基因功能分析的基礎。
基因作為生物遺傳的基本功能單元,其在各種生物體細胞內廣泛存在,且不同物種的基因由於物種進化的關係,存在著廣泛的相似和同源性。基於此,為了確定轉錄組拼接得到的基因種類,最常用的基因注釋方法就是將序列比對到已知的物種的核酸和蛋白序列,根據同源比對的方法,通過已知的同源序列的功能推測未知的序列的種類。但是這種基因注釋只能確定序列的功能,還不能對序列的結構,特別是5』和3』非編碼區(Un-Translated Region,5』-,3』-UTR)和編碼區(CoDing Sequences,CDS)進行準確地確定;而UTR和CDS的確定對於基因功能的研究非常重要,因為很多基因的調控就是通過UTR區域實現的。比如想要了解某個基因受到miRNA的調控,最直接的一個方法就是將基因的UTR序列與該物種的miRNA的種子區域進行比對。另外的一個應用就是對突變位點的生物功能的確定,就序列明確基因編碼蛋白的方式、起始和終止坐標,才能判斷該突變是不是在編碼區,會不會導致蛋白質的變化等等。
對於大量的非模式生物而言,沒有任何關於該物種的公共的相關基因序列信息,其轉錄組拼接的基因的結構分析更加困難。針對這種情況,目前基因結構分析的比較常用的方法之一就是最長編碼算法,比如NCBI的ORFFinder程序。該算法自動尋找使用者提供的核酸序列的6中編碼方式中,尋找起始密碼子和終止密碼子,找到最長的可編碼基因為其最可能編碼的序列。這個方法最大的特點是能夠找到的最長的翻譯的蛋白,運行迅速,但是該方法不能對翻譯的蛋白的功能做任何保證,並且最長的編碼也不一定就是基因編碼的蛋白序列,所以該方法往往具有較高的假陽性。為了克服最長編碼算法的假陽性,生物信息學家們提出使用馬爾科夫鏈的方法對基因的編碼方式進行確認。其主要的思路是利用編碼最長的蛋白的前100-500條基因序列作為訓練集,訓練該物種的核酸編碼蛋白的馬爾科夫鏈模型,然後利用該模型對該物種的所有核酸序列的6種編碼方式計算其概率,概率最高的為最可能的蛋白編碼方式,在確定該編碼方式下的起始密碼子和終止密碼子,從而對大量的基因序列進行結構分析。這種方法考慮到了物種特異性,使用物種的蛋白編碼的基因序列構建模型,在一定程度上提高了預測的準確度,但是這種方法仍然不能保證預測的翻譯的蛋白的功能。
技術實現要素:
本發明的目的在於提供一種能大幅提高基因編碼方式的預測準確度,並能保證大部分基因序列翻譯的蛋白質功能的非模式生物轉錄組基因序列結構分析的方法,以解決上述背景技術中提出的現有的大規模基因序列結構分析方法假陽性高,且無法保證翻譯的蛋白序列功能的問題。
為實現上述目的,本發明提供如下技術方案:
一種非模式生物轉錄組基因序列結構分析的方法,包括以下步驟:
(1)通過序列比對,得到轉錄組基因序列在公共蛋白資料庫的最優比對結果;
(2)根據比對結果,確定有比對結果的基因序列的蛋白編碼模式,確定翻譯終止位置;
(3)利用公共的蛋白序列,通過馬爾科夫鏈訓練獲得編碼起始的序列特徵,確定基因序列的編碼起始位置;
(4)使用已知的編碼蛋白的核酸序列,利用支持向量機SVM訓練編碼蛋白的基因模型,對於沒有比對上任何已知蛋白序列的基因利用上述模型進行分類;
(5)使用轉錄組序列中確定編碼方式的核酸序列,使用馬爾科夫鏈訓練編碼蛋白的核酸序列模型;
(6)對於通過SVM分類為蛋白編碼的核酸序列,通過上述馬爾科夫鏈模型,確定剩餘蛋白編碼序列的編碼方式。
作為本發明進一步的方案:步驟(1)中以公共蛋白質資料庫為參考,使用blastx程序將轉錄組拼接得到的核酸序列比對到蛋白資料庫中;對於每個核酸序列,只保留其最佳的比對結果:E值最小的比對。
作為本發明再進一步的方案:步驟(2)中將蛋白比對的結果轉換為核酸的比對,確定核酸翻譯蛋白序列的編碼方式,並按照這個編碼方式向後繼續讀取核酸三聯體密碼子,一直讀到終止密碼子結束;如果沒有讀到終止密碼,則表示該cDNA序列不完整,核酸序列全部翻譯直到序列結束;如果讀到終止密碼,則後續的序列是該基因的3』-UTR。
作為本發明再進一步的方案:步驟(3)中利用所述述公共蛋白序列庫,分別利用馬爾科夫鏈訓練起始胺基酸甲硫氨酸Met後續的胺基酸序列和非起始甲硫氨酸後續的胺基酸序列模型;利用步驟(2)中獲得的核酸編碼方式,向前提取三聯體密碼子,如果碰到Met,則利用Met後的核酸序列判斷該Met為起始密碼子的可能性。
作為本發明再進一步的方案:步驟(4)中利用步驟(3)中獲得的編碼蛋白的序列,利用多種基因序列和表達量特徵構建蛋白編碼基因的SVM模型,對於步驟(1)中未比對上任何蛋白的核酸序列,使用SVM模型模型進行分類,顯著判定為蛋白編碼的序列進行步驟(5)的分析。
作為本發明再進一步的方案:步驟(5)中利用步驟(3)中獲得的核酸編碼的馬爾科夫模型,對步驟(4)中判定為蛋白編碼的核酸序列預測最有可能的蛋白編碼開放閱讀框模式,並分別向前向後尋找起始和終止密碼子;對於Met是否為起始密碼子的確定,使用步驟(3)中的方法進行判斷。
與現有技術相比,本發明的有益效果是:本發明提供了一種生物轉錄組基因序列結構分析的方法,能夠對任何非模式生物的轉錄組測序獲得的大量的基因序列進行高通量結構分析。相對於現有的分析技術,該方法具有以下幾個優點:
(1) 不受研究物種的限制,只要相關其他物種的蛋白序列支持,就可以對任何物種的轉錄組基因序列進行結構分析;
(2)該分析過程自動完成了轉錄組序列的注釋,可以與現有的轉錄組注釋流程整合,豐富現有的轉錄組拼接得到的核酸序列功能注釋的內涵;
(3)由於本發明的方法基於的是核酸序列的公共蛋白資料庫比對,因而獲得的蛋白序列大部分都是具有明確生物學功能的胺基酸序列;
(4)本發明的方法是利用基於比對的高度可靠的蛋白編碼核酸序列構建了馬爾科夫模型和支持向量機模型,相對於其他的方法其構建模型的基礎序列數據的可信度更高;
(5) 本發明的方法對於沒有任何比對的核酸序列也應用了支持向量機對其蛋白編碼性進行預測,為物種特異基因的後續研究提供了重要的序列信息。
附圖說明
圖1為非模式生物轉錄組基因序列結構分析的方法的分析流程示意圖。
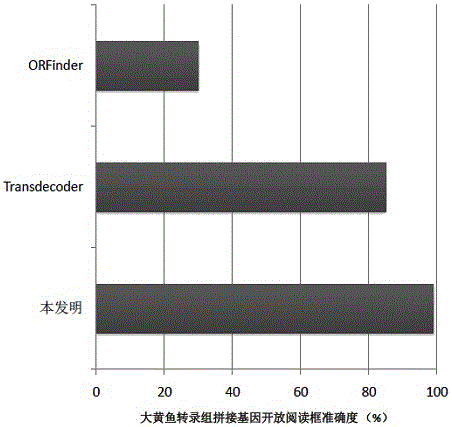
圖2為非模式生物轉錄組基因序列結構分析的方法中中大黃魚轉錄組拼接的基因序列結構分析結果比較圖。
具體實施方式
下面將結合本發明實施例中的附圖,對本發明實施例中的技術方案進行清楚、完整地描述,顯然,所描述的實施例僅僅是本發明一部分實施例,而不是全部的實施例。基於本發明中的實施例,本領域普通技術人員在沒有做出創造性勞動前提下所獲得的所有其他實施例,都屬於本發明保護的範圍。
實施例1
請參閱圖1~2,本實施例利用該發明提供的一種非模式生物轉錄組基因序列結構分析的方法,對大黃魚的轉錄組拼接產物進行基因序列結構分析。
大黃魚(Larimichthys crocea),俗稱黃魚、黃花魚,隸屬硬骨魚綱鱸形目石首魚科黃魚屬,是我國近海重要經濟魚類,有「海水國魚」之稱。目前,大黃魚是我國育苗和養殖量最多的海水魚類之一,年產量已經超過12萬噸,每年的直接經濟產值數十億元。對大黃魚的基因序列進行全面分析,是大黃魚遺傳研究重要的遺傳資源,是探討生長速度,肌肉品質和抗病性等重要經濟性狀性狀遺傳基因的基礎,也為後續進行分子輔助育種和全基因組關聯分析的提供了重要的依據。本實例利用本發明提供的非模式生物轉錄組基因序列結構分析的方法,對大黃魚轉錄組測序拼接得到的37511條基因序列進行分析,以解釋本發明的具體分析步驟和方法。為了檢驗為了說明本方法的準確度,本實例使用人的公共參考蛋白序列進行分析,包括以下步驟:
(1) 通過公共蛋白資料庫序列的局部比對,得到轉錄組基因序列的最優比對結果。在ensembl公共資料庫下載斑馬魚的全長參考蛋白序列(版本號GRCz10)。使用blast+軟體包(版本號2.4.0)的makeblastdb使用人的蛋白數據構建搜索資料庫庫,並使用blastx程序將大黃魚的37511條基因序列比對到人的蛋白資料庫上。比對的主要參數如下:-evalue 1e-5 –num_threads 32。按照E值對每一條大黃魚基因序列選擇最佳的目標序列,得到32135條序列的最佳比對;
(2)根據序列比對確定基因序列的蛋白編碼模式,確定翻譯終止位置。使用步驟(1)中的比對結果,確定有比對結果的大黃魚基因序列翻譯蛋白的開放閱讀框的編碼方式,並在大黃魚基因組序列上向後延伸三聯體密碼。32135條有比對的大黃魚序列中,有21591條成功找到終止密碼子序列,確定翻譯終止位置,其後的序列為這些基因的3』-UTR;剩餘的10544條序列沒有找到終止密碼,則一直翻譯到序列末;
(3)利用人的蛋白序列數據,通過馬爾科夫鏈訓練獲得編碼起始的序列特徵,確定基因序列的編碼起始位置。在人的蛋白序列資料庫中,分別確定Met為起始胺基酸和非起始胺基酸的後續序列,並分別利用馬爾科夫鏈構建起始Met和非起始Met的模型。在步驟(2)中有比對的序列中,向前延伸三聯體密碼子,尋找起始密碼子(ATG)。如果發現密碼子翻譯Met,則使用上述馬爾科夫模型判斷該Met是否為起始胺基酸。如果按照Met為起始胺基酸的模型計算的概率較高,則認為該ATG為翻譯起始位點,否則則繼續向前延伸蛋白序列,直到找到翻譯起始位點或者序列結束。按照這個方法,32135條有比對的大黃魚序列中,19856條序列成功找到翻譯起始位點;
(4)使用已知的編碼蛋白的核酸序列,利用支持向量機(SVM)訓練編碼蛋白的基因模型,對於沒有比對上任何已知蛋白序列的基因利用上述模型進行分類。為了進一步對為比對上任何人類蛋白的基因進行分析,利用步驟(2)和(3)中找到的32135條編碼蛋白的大黃魚序列構建SVM模型。使用的基因序列屬性包括:序列長度,GC含量,CPAT軟體預測分數,基因表達量(FPKM)。使用22135條序列進行模型訓練,10000條序列進行模型驗證,發現該SVM模型的準確度高達98.3%。對步驟1中未比對上任何人蛋白序列的5376條大黃魚基因序列,使用該SVM模型進行預測,發現其中3290條序列為蛋白編碼序列。
(5)對於步驟(4)中通過SVM分類為蛋白編碼的3290條核酸序列,利用步驟(2)的方法和步驟(3)中構建的馬爾科夫鏈模型,確定這些蛋白編碼序列的編碼方式。
(6)對於上述5步中獲得的大黃魚轉錄組序列可翻譯蛋白序列的編碼方式,利用大黃魚全基因組注釋信息提供的蛋白序列進行一一比對,驗證蛋白編碼方式檢測的準確性。並分別使用ORFinder和transdecoder進行分析,比較蛋白編碼方式判斷的準確度。
對於本領域技術人員而言,顯然本發明不限於上述示範性實施例的細節,而且在不背離本發明的精神或基本特徵的情況下,能夠以其他的具體形式實現本發明。因此,無論從哪一點來看,均應將實施例看作是示範性的,而且是非限制性的,本發明的範圍由所附權利要求而不是上述說明限定,因此旨在將落在權利要求的等同要件的含義和範圍內的所有變化囊括在本發明內。不應將權利要求中的任何附圖標記視為限制所涉及的權利要求。
此外,應當理解,雖然本說明書按照實施方式加以描述,但並非每個實施方式僅包含一個獨立的技術方案,說明書的這種敘述方式僅僅是為清楚起見,本領域技術人員應當將說明書作為一個整體,各實施例中的技術方案也可以經適當組合,形成本領域技術人員可以理解的其他實施方式。
