基於sanger測序測重組HPV腺病毒基因組的方法與流程
2023-12-01 00:39:16 3
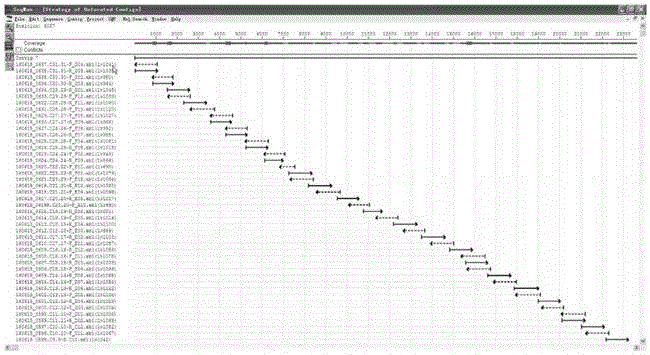
本發明涉及基因工程領域,具體涉及一種基於sanger測序測重組HPV腺病毒基因組的方法。
背景技術:
:宮頸癌是女性生殖系統常見的惡性腫瘤,研究顯示90%以上的宮頸癌與人乳頭瘤病毒的感染有關,其中超過2/3的宮頸癌與HPV16或l8感染相關,約50%是人乳頭瘤病毒16型(HPV16)感染引起的。HPV具有嚴格的嗜人類組織的特性,自然狀態下不感染任何實驗動物,這就給研究HPV的感染過程及致病機理帶來了不便。腺病毒可跨越種屬特性,並且在不同細胞、組織中都可以表達。使HPV感染更類似於臨床宮頸癌的發生過程。這對於構建HPV-16動物模型以及開發治療HPV感染的藥物等也有著重要的作用。腺病毒在生物醫藥和基因治療中因其具有以下優點,被廣泛用作於載體:外源基因容量大,最大承載可達37kb;感染範圍廣,能將目的基因轉移到分裂或靜息的細胞中;安全性高,感染細胞時其DNA不整合到宿主染色體中,潛在的致癌危險小;能同時表達多個基因,它是第1個可以在同一細胞株或組織中用來設計表達多個基因的表達系統;外源基因表達水平較高,病毒滴度高,易於製備和純化。腺病毒載體的安全性問題主要在於外源基因插入片段序列是否正確以及該片段在病毒連續複製幾個周期後是否仍保持不變。目前的檢測方法都是基於PCR擴增或者基因組酶切圖譜鑑定,但是這些方法只能定性評估重組腺病毒載體構建成功與否,沒有精確到鹼基序列的完全匹配,實際應用過程中是可能存在安全風險的。本文提供了一種基於sanger測序法測重組HPV腺病毒基因組的方法,為滿足疫苗研發對病毒種子的要求,以及下一步的中試研發提供了技術保障。技術實現要素:本發明的目的在於克服上述現有技術存在的不足,提供一種基於sanger測序測重組HPV腺病毒基因組的方法。本發明的目的是通過以下技術方案來實現的:一種基於sanger測序測重組HPV腺病毒基因組的方法,所述方法包括如下步驟:S1、病毒DNA提取;S2、根據NCBI資料庫中已知的重組HPV腺病毒序列設計覆蓋線粒體全長的引物對,對病毒DNA進行PCR擴增;S3、回收PCR擴增產物;S4、對所述回收產物進行測序;用Seqman序列拼接軟體對測得的序列根據引物對與引物對之間的序列重疊區進行拼接,組裝得到重組HPV腺病毒基因組全序列。優選的,步驟S2中,所述引物對包括:如SEQIDNO.1所示的1-F和如SEQIDNO.2所示的1-R構成的引物對;如SEQIDNO.3所示的2-F和如SEQIDNO.4所示的2-R構成的引物對;如SEQIDNO.5所示的3-F和如SEQIDNO.6所示的3-R構成的引物對;如SEQIDNO.7所示的4-F和如SEQIDNO.8所示的4-R構成的引物對;如SEQIDNO.9所示的5-F和如SEQIDNO.10所示的5-R構成的引物對;如SEQIDNO.11所示的6-F和如SEQIDNO.12所示的6-R構成的引物對;如SEQIDNO.13所示的7-F和如SEQIDNO.14所示的7-R構成的引物對;如SEQIDNO.15所示的8-F和如SEQIDNO.16所示的8-R構成的引物對;如SEQIDNO.17所示的9-F和如SEQIDNO.28所示的9-R構成的引物對;如SEQIDNO.19所示的10-F和如SEQIDNO.20所示的10-R構成的引物對;如SEQIDNO.21所示的11-F和如SEQIDNO.22所示的11-R構成的引物對;如SEQIDNO.23所示的12-F和如SEQIDNO.24所示的12-R構成的引物對;如SEQIDNO.25所示的13-F和如SEQIDNO.26所示的13-R構成的引物對;如SEQIDNO.27所示的14-F和如SEQIDNO.28所示的14-R構成的引物對;如SEQIDNO.29所示的15-F和如SEQID.NO.30所示的15-R構成的引物對;如SEQIDNO.31所示的16-F和如SEQIDNO.32所示的16-R構成的引物對;如SEQIDNO.33所示的17-F和如SEQID.NO.34所示的17-R構成的引物對;如SEQIDNO.35所示的18-F和如SEQIDNO.36所示的18-R構成的引物對;如SEQIDNO.37所示的19-F和如SEQIDNO.38所示的19-R構成的引物對;如SEQIDNO.39所示的20-F和如SEQIDNO.40所示的20-R構成的引物對;如SEQIDNO.41所示的21-F和如SEQIDNO.42所示的21-R構成的引物對;如SEQIDNO.43所示的22-F和如SEQIDNO.44所示的22-R構成的引物對;如SEQIDNO.45所示的23-F和如SEQIDNO.46所示的23-R構成的引物對;如SEQIDNO.47所示的24-F和如SEQIDNO.48所示的24-R構成的引物對;如SEQIDNO.49所示的25-F和如SEQIDNO.50所示的25-R構成的引物對;如SEQIDNO.51所示的26-F和如SEQIDNO.52所示的26-R構成的引物對;如SEQIDNO.53所示的27-F和如SEQIDNO.54所示的27-R構成的引物對;如SEQIDNO.55所示的28-F和如SEQIDNO.56所示的28-R構成的引物對;如SEQIDNO.57所示的29-F和如SEQIDNO.58所示的29-R構成的引物對;如SEQIDNO.59所示的30-F和如SEQIDNO.60所示的30-R構成的引物對;如SEQIDNO.61所示的31-F和如SEQIDNO.62所示的31-R構成的引物對;如SEQIDNO.63所示的32-F和如SEQIDNO.64所示的32-R構成的引物對;優選的,步驟S2中,所述PCR擴增中,每50.0ul擴增反應體系包括:基因組DNA1.0ul,含2.5mMMg2+的10×LABuffer5.0ul,5u/μL的LATaq聚合酶1.0ul,10mM的dNTP1.0ul,10uM的正向引物1.5ul,10uM的反向引物1.5ul,ddH2O39.0ul。優選的,步驟S2中,所述PCR擴增中,PCR反應參數為:預變性95℃,5min;變性95℃,30s;退火58℃,30s;延伸72℃,1min30s;終延伸72℃,7m;循環數35。優選的,步驟S3中,所述回收是用AxyPrepDNA凝膠回收試劑盒進行回收。優選的,步驟S4中,所述測序是基於Sanger雙脫氧鏈終止法進行一代測序。該方法的基本原理是通過已知的重組HPV腺病毒序列設計出覆蓋基因組全長的引物對,分段擴增出特定的基因片段,然後用ABI3730xl測序儀測出這些基因的序列,然後通過序列與序列之間的重疊區將這些序列拼接成一個完整的重組HPV腺病毒載體。與現有技術相比,本發明具有如下有益效果:本發明通過對重組腺病毒載體的全基因組測序,從鹼基序列上保證了重組質粒的準確性,進而確保了疫苗生產的安全性問題。附圖說明圖1為測得的序列根據引物對與引物對之間的序列重疊區進行拼接的效果示意圖。具體實施方式下面結合實施例對本發明進行詳細說明。以下實施例將有助於本領域的技術人員進一步理解本發明,但不以任何形式限制本發明。應當指出的是,對本領域的普通技術人員來說,在不脫離本發明構思的前提下,還可以做出若干調整和改進。這些都屬於本發明的保護範圍。實施例本實施例涉及一種基於sanger測序測重組HPV腺病毒基因組的方法;具體包括如下步驟:一、病毒DNA提取1、取病毒上清液0.5ml,12000rpm離心5min,儘量吸盡上清使用,棄去沉澱。2、向病毒上清中加入20ul10mg/ml的蛋白酶K,充分混勻,65℃消化10-20min,期間可顛倒離心管混勻數次。3、向管中加入500ul結合液,充分混勻。再向管中加入400ul無水乙醇,充分混勻,此時可能會出現絮狀沉澱,不影響DNA的提取,可將溶液和絮狀沉澱都加入吸附柱中,靜置2min。(吸附柱的最大容積為750ul,可分兩次加入。一次吸附完離心後再將餘下的混合液體加入柱中靜置離心。)4、12000rpm離心2min,棄廢液,將吸附柱放入收集管中。5、向吸附柱中加入700ul漂洗液(使用前請先檢查是否已加入無水乙醇),12000rpm離心1min,棄廢液,將吸附柱放入收集管中。6、向吸附柱中加入500ul漂洗液,12000rpm離心1min,棄廢液,將吸附柱放入收集管中。7、12000rpm離心2min,將吸附柱置於室溫或50℃溫箱放置數分鐘,目的是將吸附柱中殘餘的漂洗液去除,否則漂洗液中的乙醇會影響後續的實驗如酶切、PCR等。8、將吸附柱放入一個乾淨的離心管中,向吸附膜中央懸空滴加50ul-100ul經65℃水浴預熱的洗脫液,室溫放置5min,12000rpm離心1min。9、離心所得洗脫液再加入吸附柱中,室溫放置2min,12000rpm離心2min,即可得到高質量的病毒基因組二、引物設計與合成根據NCBI資料庫中已知的重組HPV腺病毒序列設計覆蓋線粒體全長的引物對,對病毒DNA進行PCR擴增,這些引物由上海派森諾生物科技有限公司合成,引物序列如下:三、PCR擴增:對總DNA進行擴增,得到特定的基因片段3.1PCR擴增反應體系表1基因組DNA1.0ul10×LABuffer(含2.5mMMg2+)5.0ulLATaq聚合酶(5u/μL)1.0uldNTP(10mM)1.0ul正向引物(10uM)1.5ul反向引物(10uM)1.5ulddH2O39.0ul總體積50.0ul在0.2ml離心管中加入表1所示成分,混勻,瞬時離心收集管壁上的液滴至管底,在PCR擴增儀上進行PCR反應,反應參數如下表2所示:表23.2PCR產物的回收PCR產物用AxyPrepDNA凝膠回收試劑盒回收,具體操作按試劑盒說明書進行,步驟如下:3.2.1在紫外燈下切下含有目的DNA的瓊脂糖凝膠放入乾淨的離心管中,稱取重量。3.2.2加入3個凝膠體積的BufferDE-A,混合均勻後於75℃加熱直至凝膠塊完全熔化。3.2.3加0.5個BufferDE-A體積的BufferDE-B,混合均勻;當分離的DNA片段小於400bp時,加入1個凝膠體積的異丙醇。3.2.4將混合液,轉移到DNA製備管12,000×g離心1min。棄濾液。3.2.5將製備管置回2ml離心管,加500μlBufferW1,12,000×g離心30s,棄濾液。3.2.6將製備管置回2ml離心管,加700μlBufferW2,12,000×g離心30s,棄濾液。以同樣的方法再用700μlBufferW2,12,000×g離心1min。3.2.7將製備管置回2ml離心管中,12,000×g離心1min。3.2.8將製備管置於潔淨的1.5ml離心管(試劑盒內提供)中,在製備膜中央加25-30μl去離子水,室溫靜置1min。12,000×g離心1min洗脫DNA。四、一代測序利用基於Sanger雙脫氧鏈終止法的技術進行一代測序,所用測序儀為ABI3730xl。五、序列拼接用Seqman軟體對測得的序列根據引物對與引物對之間的序列重疊區進行拼接,拼接效果如圖1所示,由圖1可知,將測得的序列根據引物對與引物對之間的序列重疊區進行拼接,得到完整的基因組序列。當前第1頁1 2 3 
