草魚呼腸孤病毒的核酸適配體及其衍生物、篩選方法和應用與流程
2023-08-01 00:38:01 2
本發明屬於分子生物學領域,更具體地,涉及草魚呼腸孤病毒的核酸適配體及其衍生物、篩選方法和應用。
背景技術:
草魚呼腸孤病毒(grasscarpreovirus,gcrv)被公認為是水生呼腸孤病毒中致病力最強的毒株,引起草魚出血病,造成魚體大量死亡,死亡率高達85%以上。迄今為止,魚體一旦被gcrv感染,沒有有效的抗病毒治療方法。雖然疫苗可以有效的預防該病,但是近年來不斷出現病毒變異株,單一化的疫苗使用受到了限制。因此,這些疾病的預防在草魚養殖中仍然是一個巨大的挑戰。
核酸適配體是一種可以特異性結合靶標的dna或者rna寡核苷酸;由於其分子量較小,可化學合成、穩定性好、沒有毒性,與靶標親和力高等優點,有望應用於分子檢測領域。此外,只有少部分核酸適配體具有阻斷病毒與宿主細胞結合併抑制病毒感染的能力,這些具有抗病毒能力的核酸適配體在病毒性疾病治療中有非常廣闊的前景。
然而,核酸適配體經過了二十多年的發展,至今並沒有得到廣泛的應用,其原因在於:核酸適配體的篩選需要從一個高容量的隨機庫中篩選出針對靶標的適配體,其篩選過程十分困難,要獲得某種靶標的適配體,往往需要不斷的摸索和創造,才能獲得的具有良好應用價值的核酸適配體序列。
技術實現要素:
本發明的目的在於提供草魚呼腸孤病毒的核酸適配體及其衍生物、篩選方法和應用。
本發明所採取的技術方案是:
草魚呼腸孤病毒的核酸適配體,其核苷酸序列為下述seqidno:1~10所示序列的任意一條序列:
ft1:5』-catttttcctttgcttgccgagctccagtgtatca-3』(seqidno:1);
ft2:5』-aattgtctgttacctctcggcactctaacacaatg-3』(seqidno:2);
ft3:5』-gattgtgcaacgttcctgtcgtgttgtccatagat-3』(seqidno:3);
ft4:5』-ttcttattgacttttgggtgaatgtctcttatat-3』(seqidno:4);
ft5:5』-tcatagtgggcggtctttgcagtcgttactctcca-3』(seqidno:5);
ft6:5』-cattgtgttatacgagaatgactccgttaaaatt-3』(seqidno:6);
ft7:5』-gacagtatactatattttttctaaatgtttctttt-3』(seqidno:7);
ft8:5』-taccgagtggtagaatttgcttggattatttttta-3』(seqidno:8);
ft9:5』-gattgtgcaacgttcctgtcgtgttgtccatagat-3』(seqidno:9);
ft10:5』-tttttaaatttttgttcttattcctatcttcaaa-3』(seqidno:10)。
核酸適配體,其包含以下三種序列中的任意一種:
(1)與seqidno:1~10所示的核酸適配體序列的序列同源性在60%以上的序列;
(2)能夠與seqidno:1~10所示的核酸適配體的序列進行雜交的序列;
(3)由seqidno:1~10所示的核酸適配體的序列反轉錄的序列。
核酸適配體衍生物,所述衍生物是以上所述的核酸適配體的1個或多個核苷酸進行衍生獲得:衍生的方式為下述修飾或化學改造或連接標記物中的一種或多種:聚乙二醇修飾、鎖核酸修飾、磷酸化修飾;甲基化修飾;氨基化修飾、巰基化修飾、同位素化修飾、連接螢光標記物、連接放射性物質、連接治療性物質、連接生物素、連接地高辛、連接納米發光材料、連接酶標記。
如上所述的核酸適配體或核酸適配體衍生物在製備預防和/或治療草魚呼腸孤病毒感染藥物中的應用。
如上所述的核酸適配體或核酸適配體衍生物在製備草魚呼腸孤病毒分子探針、檢測試劑或靶向介質中的應用。
草魚呼腸孤病毒的核酸適配體的篩選方法,其特徵在於:包括如下步驟:
(1)合成ssdna隨機適配體庫和pcr擴增引物:
ssdna隨機適配體庫:
5』-atccagagtgacgcagca-(35n)-actaagccaccgtgtcca-3』;
pcr擴增引物:
正向引物:5』-atccagagtgacgcagca-3』;
反向引物:5』-actaagccaccgtgtcca-3』,所述反向引物5』端用生物素標記;
(2)病毒純化:
分別將草魚呼腸孤病毒感染的fhm細胞及未感染的fhm細胞,進行反覆凍融和梯度離心,分別獲得草魚呼腸孤病毒液和fhm細胞液,備用;
(3)selex篩選:
正向篩選:
將步驟(1)中的ssdna隨機適配體庫進行變性處理,隨後快速置於冰上放置8~15min,加入到綁定有步驟(2)草魚呼腸孤病毒液的96孔板中,孵育50~70min;用緩衝液洗脫2~4次,去除未綁定序列,再進行變性處理,回收綁定的序列;
以綁定的序列作為模板,採用步驟(1)中的引物,進行pcr擴增,得到ssdna文庫;
將ssdna文庫替代前述的ssdna隨機適配體庫,進行下一輪篩選;
當正向篩選重複4~5輪後,進入反向篩選;
反向篩選:
將上一輪正向篩選獲得的ssdna文庫加入到綁定有步驟(2)fhm細胞液的96孔板中,孵育50~70min;回收未綁定序列,用於替代正向篩選步驟中的ssdna隨機適配體庫,再重複正向篩選;
重複篩選至獲得草魚呼腸孤病毒的核酸適配體序列,終止篩選。
作為上述篩選方法的優選,步驟(3)中,隨篩選輪數的增加,孵育溫度由2~6℃逐步提高至26~30℃。
作為上述篩選方法的優選,步驟(2)中,反覆凍融條件為:–85~–75℃反覆凍融2~4次,梯度離心條件為2500~3500rpm/min,13~17min;4500~5500rpm/min,13~17min;7500~8500rpm/min,18~22min;9500~10500rpm/min,28~32min。
作為上述篩選方法的優選,變性處理的條件是94~96℃作用5~10min。
作為上述篩選方法的優選,pcr擴增反應條件為:94℃5min,94℃30s,53℃30s,72℃20s,16個循環,72℃7min。
本發明的有益效果是:
本發明的草魚呼腸孤病毒的核酸適配體是針對gcrv病毒的ssdna庫,應用selex方法獲得的核酸適配體,採取了純化的病毒,逐步提高孵育溫度,增加反向篩選等技術改進使針對靶標的序列更容易富集,最終篩選到針對gcrv的適配體,其核苷酸序列為seqidno:1~10所示的任意一條序列,經過實驗表明:以上核酸適配體對細胞活力沒有明顯影響,能形成莖-環的二級結構,甚至可以抑制gcrv的感染,其中ft6和ft7適配體的抗病毒效果最佳,其形成的都是從約十三個寡核苷酸到第二十七個寡核苷酸的莖環。
本發明的草魚呼腸孤病毒的核酸適配體可特異性的抑制抑制gcrv感染,具有製備預防和治療草魚呼腸孤病毒感染藥物的應用前景,同時,根據適配體的自身特性,也具有製備草魚呼腸孤病毒分子探針、檢測試劑或靶向介質的應用前景。
附圖說明
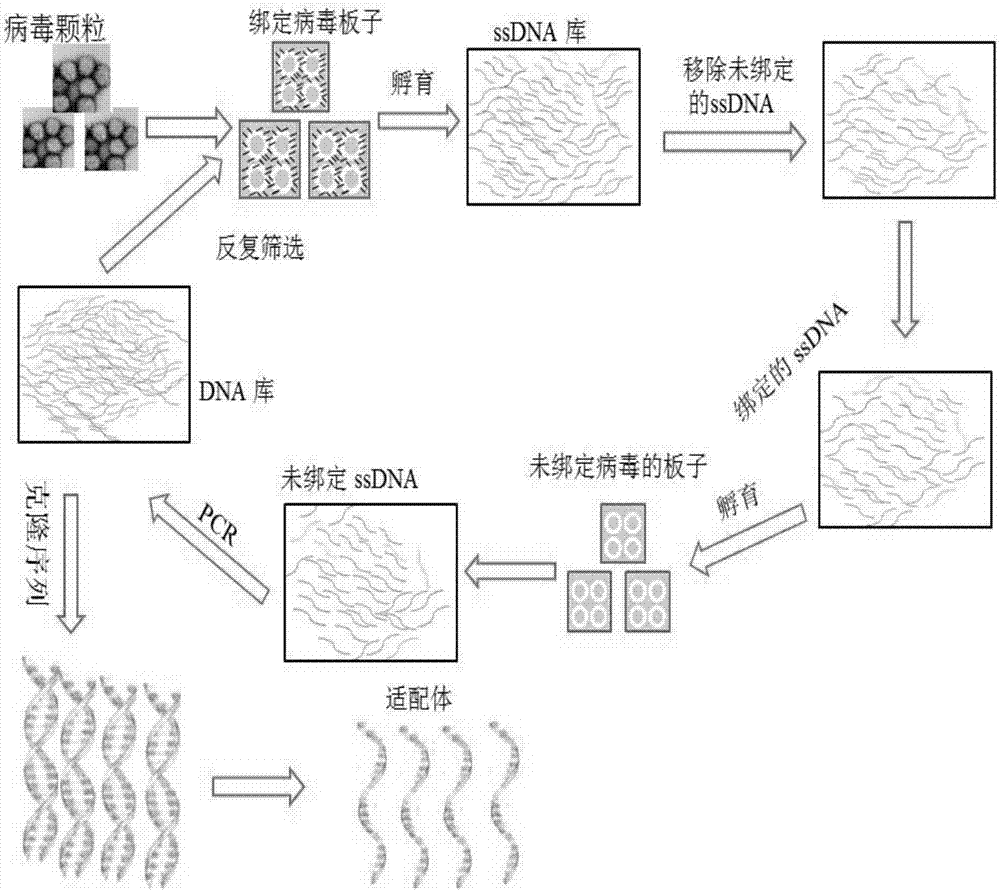
圖1:selex技術示意圖;
圖2:核酸適配體的二級結構圖,圖a為ft1~5核酸適配體的二級結構,圖b為ft6~10核酸適配體的二級結構;
圖3:實施例3的mtt法檢測細胞活力結果;
圖4:核酸適配體對gcrv病毒的抑制作用,圖a為核酸適配體和gcrv同時加入細胞;圖b為gcrv感染1小時後加入核酸適配體;圖c為核酸適配體提前加入細胞,1小時後gcrv感染,*表示p<0.05。
具體實施方式
本發明中,所述的核酸適配體序列可選自天然存在或人工合成的序列,或任何其他來源的同樣的序列。
本發明中,所述的可以為:
(1)seqidno:1~10所示的任意一條序列;
(2)與seqidno:1~10所示的任意一條序列的序列同源性在60%以上的序列;
(3)能夠與seqidno:1~10所示的任意一條序列進行雜交的序列;
(4)由seqidno:1~10所示的任意一條序列反轉錄的序列。
本發明中,核酸適配體衍生物是通過以上核酸適配體序列的1個或多個核苷酸進行衍生獲得:所述衍生的方式為下述修飾或化學改造或連接標記物中的一種或多種:聚乙二醇(peg)修飾、鎖核酸修飾、磷酸化修飾;甲基化修飾;氨基化修飾、巰基化修飾、同位素化修飾、連接螢光標記物、連接放射性物質、連接治療性物質、連接生物素、連接地高辛、連接納米發光材料、連接酶標記。其中,peg修飾可以克服核酸適配體在應用中存在一些弊端,如核酸適配體由於分子量太小而易被酶降解;在機體內也容易被巨噬細胞吞噬清除,半衰期短,不利於適配體的後期應用的弊端。本發明通過peg5000對適配體進行修飾,提高了篩選適配體的穩定性,利於適配體後期應用與研究。
本發明的selex篩選在每輪篩選中,逐步提高孵育溫度,從2~6℃逐步提高到26~30℃,其目的是在低溫下利於形成結構和靶標結合,逐步提高溫度是增加篩選條件的苛刻性,使特異性的適配體留下,非特異性的排除。
以下實施例是對本發明的進一步說明,而不是對本發明的限制。
以下實施例中的實驗方法如無特殊說明,均為常規方法。以下實施例中的實驗材料如無特殊說明,均是從常規的生化試劑公司購買所得到。
本發明所用的gcrv毒株和胖頭鱥肌肉細胞(fhm細胞)由珠江水產研究所魚病室提供,其中,fhm細胞28℃培養於含10%的胎牛血清m199培養基;gcrv毒株置於–80℃備用。
實施例1、草魚呼腸孤病毒的核酸適配體的篩選
利用指數富集配體系統進化技術(selex技術)進行草魚呼腸孤病毒的核酸適配體的篩選,selex技術示意圖參見圖1,具體實施方法如下:
(1)病毒純化
將gcrv接種於fhm細胞置於28℃培養直至80%左右出現病變收穫,收穫後於–80℃反覆凍融3次,細胞碎片按梯度離心除去:3000rpm/min,15min;5000rpm/min,15min;8000rpm/min,20min;10000rpm/min,30min,獲得gcrv病毒液。同時未感染的fhm細胞作為對照,用於篩選過程的反向篩選,也進行相同條件反覆凍融和梯度離心的處理,獲得fhm細胞液,–80℃保存備用。
(2)合成selex篩選的庫和引物
20nmol的ssdna隨機適配體庫:中間為35個鹼基的隨機序列,兩端各為18個鹼基的固定序列:5′-atccagagtgacgcagca-3′和5′-actaagccaccgtgtcca-3′;兩端序列用於pcr擴增,pcr擴增引物為:正向引物:5』-atccagagtgacgcagca-3』(seqidno:11)和反向引物:5』-actaagccaccgtgtcca-3』(seqidno:12),反向引物5′端用生物素標記。
(3)selex篩選
將步驟(2)的gcrv病毒液按100ul/孔加入96孔板4℃,過夜,然後細胞用pbs洗脫3次,除去未綁定的序列,獲得綁定有純化的gcrv病毒的96孔板;將步驟(1)ssdna隨機適配體庫於95℃作用5min使其變性,然後快速置於冰上放置10min,加入到綁定有gcrv病毒液的96孔板中,4℃孵育60min;用pbs洗脫96孔板3次,除去未綁定的序列,然後96孔板於95℃作用10min,使綁定序列解離,回收綁定的序列。
綁定的序列作為模板進行pcr擴增,採用步驟(1)所示的pcr擴增引物,按反應條件擴增:94℃5min,94℃30s,53℃30s,72℃20s,16個循環,72℃7min,分離獲得ssdna文庫;
將ssdna文庫替代前述的ssdna隨機適配體庫,進行下一輪篩選;
當正向篩選重複4輪後,進入反向篩選。
(4)反向篩選
將上一輪正向篩選獲得的ssdna文庫加入到綁定有步驟(2)fhm細胞液的96孔板中,4℃孵育60min;回收未綁定序列,用於替代正向篩選步驟中的ssdna隨機適配體庫,再重複正向篩選;
(5)重複篩選
前4輪為正向篩選,然後在第5輪,第10輪,第15輪進行反向篩選,篩選的溫度逐漸提高,從4℃逐漸提高到28℃,重複篩選15輪,終止篩選,將pcr純化產物連接於peasy-t1載體,轉化後挑取單菌落進行測序,測序並分析獲得適配體序列,如下所示:
ft1:5』-catttttcctttgcttgccgagctccagtgtatca-3』(seqidno:1);
ft2:5』-aattgtctgttacctctcggcactctaacacaatg-3』(seqidno:2);
ft3:5』-gattgtgcaacgttcctgtcgtgttgtccatagat-3』(seqidno:3);
ft4:5』-ttcttattgacttttgggtgaatgtctcttatat-3』(seqidno:4);
ft5:5』-tcatagtgggcggtctttgcagtcgttactctcca-3』(seqidno:5);
ft6:5』-cattgtgttatacgagaatgactccgttaaaatt-3』(seqidno:6);
ft7:5』-gacagtatactatattttttctaaatgtttctttt-3』(seqidno:7);
ft8:5』-taccgagtggtagaatttgcttggattatttttta-3』(seqidno:8);
ft9:5』-gattgtgcaacgttcctgtcgtgttgtccatagat-3』(seqidno:9);
ft10:5』-tttttaaatttttgttcttattcctatcttcaaa-3』(seqidno:10)。
實施例2、核酸適配體的空間結構預測
將上述篩選出序列進行分析以及重複出現的頻率確定的10個核酸適配體預測其空間結構:根據mfold軟體的能量原理選擇最小能量結構為適配體的最穩定結構。
結果如圖2所示,所有適配體可以形成莖-環結構,其形成的都是從約十三個寡核苷酸到第二十七個寡核苷酸的莖環,該結構可能是適配體結合靶標並發揮作用的關鍵性位點。
實施例3、核酸適配體對fhm細胞活力的影響
將fhm細胞接種於96孔板,待細胞生長至80%時,分別加入0μm至10μm的濃度的適配體ft1,28℃培養48小時,加入20μl/孔mtt溶液,28℃培養4小時,使用elisa板讀數器在490nm讀數。
實驗結果如圖3所示,核酸適配體ft1按0um到10um濃度添加到細胞後,細胞活力均沒有顯著變化,說明核酸適配體對宿主細胞不會產生毒副作用,有利於後期製備藥物及檢測試劑的應用。
實施例4、核酸適配體抑制gcrv感染實驗
(1)peg修飾
0.1m的peg5000,0.4m的1-乙基-(3-二甲基氨基丙基)碳醯二亞胺鹽酸鹽(edc.hcl)和0.1m的n-羥基琥珀醯亞胺(nhs),在水中混合併以80rpm溫育15分鐘;然後將核苷酸序列如seqidno:1~10所示的核酸適配體分別加入到混合物中,反應在80rpm下繼續進行4小時,獲得peg修飾後的每條核酸適配體,保存備用。
(2)gcrv感染實驗
將fhm細胞接種於96孔板,待生長至80%時,進行以下三組實驗:gcrv(moi=0.5)和peg修飾的核酸適配體(200nm)同時加入fhm細胞;fhm細胞gcrv(moi=0.5)感染1小時後,將peg修飾的適配體(200nm)加入培養基中;細胞與peg修飾的適配體(200nm)孵育1小時後,用gcrv(moi=0.5)感染fhm細胞。最後,將fhm細胞在28℃培養48小時,並使用reed-muench方法測定病毒滴度。
考察核酸適配體對病毒複製的抑制作用,結果如圖4所示,圖c中,當適配體提前1個小時加入到細胞,然後再加入病毒時,核酸適配體抑制病毒複製能力顯著,其中核酸適配體ft6和ft7對gcrv病毒的抑制效果顯著,說明本發明核酸適配體可用於製備預防gcrv感染的藥劑;此外,圖b顯示病毒感染1小時後加入適配體,也有一定的抑制病毒複製能力,說明核酸適配體可用於製備治療gcrv感染的藥劑。
sequencelisting
中國水產科學研究院珠江水產研究所
草魚呼腸孤病毒的核酸適配體及其衍生物、篩選方法和應用
12
patentinversion3.5
1
35
dna
人工序列
1
catttttcctttgcttgccgagctccagtgtatca35
2
35
dna
人工序列
2
aattgtctgttacctctcggcactctaacacaatg35
3
35
dna
人工序列
3
gattgtgcaacgttcctgtcgtgttgtccatagat35
4
34
dna
人工序列
4
ttcttattgacttttgggtgaatgtctcttatat34
5
35
dna
人工序列
5
tcatagtgggcggtctttgcagtcgttactctcca35
6
34
dna
人工序列
6
cattgtgttatacgagaatgactccgttaaaatt34
7
35
dna
人工序列
7
gacagtatactatattttttctaaatgtttctttt35
8
35
dna
人工序列
8
taccgagtggtagaatttgcttggattatttttta35
9
35
dna
人工序列
9
gattgtgcaacgttcctgtcgtgttgtccatagat35
10
34
dna
人工序列
10
tttttaaatttttgttcttattcctatcttcaaa34
11
18
dna
人工序列
11
atccagagtgacgcagca18
12
18
dna
人工序列
12
actaagccaccgtgtcca18
