基於相互作用指紋和機器學習的藥物靶標的虛擬篩選方法與流程
2023-05-29 11:06:11 2
本發明涉及藥物虛擬篩選技術領域。尤其是一種基於相互作用指紋和機器學習的藥物靶標的虛擬篩選方法,該方法在傳統的分子對接的基礎上,通過機器學習對已知活性及非活性小分子與靶標蛋白的相互作用指紋進行訓練得出靶標的篩選模型。
背景技術:
在新藥發現過程中,虛擬篩選的應用可以提高活性分子的富集,降低篩選的成本。近年來已引起科研機構和製藥公司的高度重視。常用的虛擬篩選方法可以分為基於結構的虛擬篩選(SBVS)和基於配體的虛擬篩選(LBVS)。基於配體的虛擬篩選的主要科研精力放在各種分子描述符的產生、相似性的比較。基於配體的虛擬篩選的優勢在於速度更快,一般可作為虛擬篩選的最初階段,劣勢在於很難找到不同於已知分子的新的骨架。基於結構的虛擬篩選雖然速度有所下降,但是可以利用靶標的信息,從而有利於全新藥物的發現。
分子對接是常用的基於結構的虛擬篩選方法。常用的分子對接軟體有:GOLD,FlexX,Glide,Fred,AutoDock,Dock等。分子對接可以分為兩步,第一步小分子以不同構象結合到結合口袋,第二步,利用打分函數進行打分。很多研究證明大部分情況下小分子可以找到合適的結合方式,但是打分函數卻存在各種問題。按照建立的順序,常用的打分函數包括:PLP,ChemScore,X-Score,and GlideScore。對接軟體中的打分函數,是根據很多已知結合能的蛋白結構利用各種相互作用擬合得到的。
機器學習已經被廣泛的應用於藥物設計的各個領域,包括靶標預測、毒性預測、藥物相似性預測、藥物活性預測等。常用的機器學習方法包括支持向量機、決策樹、貝葉斯、K鄰近和人工神經網絡等。
一方面,現有的打分函數很難考慮到不同相互作用之間的耦合作用由於數據集的局限性,另一方面對於特定蛋白來說不具有針對性。最終導致在虛擬篩選中假陽性的概率很高。因此,本領域迫切需要針對特定蛋白的篩選方法,以期提高活性分子的富集程度,提高虛擬篩選的成功率,降低虛擬篩選的成本。
技術實現要素:
本發明的目的在於提供一種基於相互作用指紋和機器學習的藥物靶標的虛擬篩選方法,以彌補現有技術的不足。
本發明的目的是這樣實現的:
一種基於相互作用指紋和機器學習的藥物靶標的虛擬篩選方法,該方法包括以下具體步驟:
步驟1:從CHEMBL、BindingDB或DUD-E資料庫或文獻中提取靶標的活性數據;
步驟2:對活性分子和非活性分子進行相似性分析,以保證活性與非活性數據的多樣性;
步驟3:分子對接,利用薛丁格分子對接軟體進行分子對接,每個小分子只保留打分最好的構象;
步驟4:計算找出結合口袋附近的胺基酸殘基;
步驟5:計算每個小分子與結合口袋中的胺基酸殘基的相互作用能,形成相互作用能矩陣;
步驟6:統計相互作用能矩陣中每個元素出現的概率,去除出現次數較少的元素,形成相互作用指紋;
步驟7:生成支持向量機輸入文件,利用網格搜索和交叉驗證尋找最優參數;
步驟8:利用步驟7得到的最優參數,交叉驗證評估模型;
步驟9:利用步驟7得到的最優參數,訓練全部樣本,得到篩選模型;
步驟10:利用篩選模型進行虛擬篩選。
所述的步驟1中,提取靶標的活性數據:要求活性小分子的IC50、Kd、Ki值小於10μM,非活性小分子從ZINC資料庫中提取。
所述的步驟2中,對活性分子和非活性分子進行相似性分析:要求活性小分子間的ECFP4相似性小於0.8;非活性小分子是以活性小分子為模板挑選的,要求兩者物理上相似但化學性質上不相似;以每個活性分子為模板,尋找與活性小分子的分子質量之差不大於20,總的重原子數之差小於2,可旋轉鍵的數目之差不大於1,氫鍵供體的數目之差不大於1,氫鍵受體的數目之差不大於2,脂水分配係數之差不大於1的小分子;非活性小分子間的相似性小於0.4,非活性小分子與每個活性小分子的ECFP4相似性小於0.6;最後得到的活性小分子的數目大於100,非活性小分子的數目是活性小分子數目的50倍。
所述的步驟4中,找出結合口袋附近的胺基酸殘基:選定靶標PDB自身配體周圍範圍內的殘基作為候選殘基,以保證可以包圍絕大部分的小分子。
所述的步驟5中,計算每個小分子與結合口袋中的胺基酸殘基的相互作用,具體是指計算對接後每個小分子與胺基酸殘基的範德華、氫鍵和疏水相互作用;三種相互作用的定義如下:
1>範德華相互作用為:
其中,i代表結合口袋中的第i個殘基,j代表配體小分子中的原子,k代表蛋白質胺基酸殘基中的原子,d0代表j原子與k原子的半徑之和,djk代表j原子與k原子的實際距離,這裡採用了8-4形式的範德華相互作用形式;
2>疏水相互作用為:
其中
其中,i代表結合口袋中的第i個殘基,j代表配體小分子中的原子,k代表蛋白質胺基酸殘基中的原子,d0代表j原子與k原子的半徑之和,djk代表j原子與k原子的實際距離;
3>氫鍵相互作用為:
要求氫鍵的供體與受體重原子之間的距離小於供體重原子、供體氫原子、受體重原子三者之間的夾角小於120度;氫鍵的大小由氫鍵供體和受體重原子之間的距離確定;
其中,i代表結合口袋中的第i個殘基,j代表配體小分子中的原子,k代表蛋白質胺基酸殘基中的原子,j與k表示氫鍵受體或供體中的重原子,djk代表j原子與k原子的實際距離。
所述的步驟5中,形成相互作用矩陣:將結合口袋中胺基酸按照從小到大的方式重新編號,每個胺基酸有範德華、疏水和氫鍵三種相互作用;形成下表所示的相互作用矩陣。
所述的步驟6中,統計相互作用能矩陣中每個元素出現的頻率:如果相互作用矩陣中某一元素出現的頻率小於0.1那麼去除該維元素。
所述的步驟7中,生成支持向量機輸入文件,利用網格搜索和交叉驗證尋找最優參數:首先需要將支持向量機軟體(libsvm)中的評價指標改為受試者工作特徵曲線下面的面積(AUC),其次考慮到數據的不平衡性,在搜索過程中w1參數設定為50,再次,設定SVM訓練的核函數為徑向基核函數(RBF)。在此基礎上利用網格搜索和交叉驗證尋找最優參數,需要搜索的參數為(C,γ),設定以下C與γ的參數組合方式:
C=2-5,2-4.5,2-4,......,215;
γ=2-15,2-14.5,2-14,......,25;
其中,C為懲罰因子,γ為RBF核參數;如果多種(C,γ)組合方式都可以使評價指標達到最優,選取C值最小的組合。
所述的步驟8中,利用交叉驗證評估模型:利用富集因子和受試者工作特徵曲線通過5折的交叉驗證對模型進行評估;具體為:
富集因子由以下公式得出
其中,As代表設定的百分位下活性分子的數目,Ds代表選定的百分位下非活性分子的數目,At代表活性分子總體的數目,Dt代表非活性分子的總體數目;公式(4)中,分子計算的是在選定的百分位中活性分子所佔的比例,分母計算的是背景分布中活性分子所佔的比例。
在實際計算中,分為以下三步:
1>計算理想情況下的富集分布。最理想的情況即全部的活性分子都排在非活性分子之前,按照此分布進行計算。
2>計算分子對接得到的結果。首先對所有對接結果進行排序,然後按照公式(4)進行計算。
3>計算SVM模型預測出來的結果。首先按照預測出來的可能性進行排序,然後按照公式(4)進行計算。
受試者工作特徵曲線,根據不同的分界值,以真陽性率為縱坐標,假陽性率為橫坐標繪製曲線。其中真陽性率為活性分子被預測為活性分子的比例,假陽性率為非活性分子被預測為活性分子的比例。定義AUC值為受試者工作特徵曲線的面積。該值越高表明假陽性的概率越低。
本發明充分利用已知活性和非活性的小分子的相互作用指紋,藉助機器學習,構建了一種更加高效的虛擬篩選方法。
本發明的有益效果
相比於傳統的方法,本發明的有益效果為:
(1)針對具體靶標進行專項訓練,可以充分考慮每種靶標的特異性,避免了傳統打分函數擬合不足的缺陷。
(2)計算每個小分子與結合口袋中每個殘基的相互作用能,有利於發現有效的結合位點或結合方式。
(3)利用機器學習進行非線性擬合,相較於線性擬合更有利於處理各個相互作用能之間的關聯或耦合作用。
(4)結果表明利用本發明,更有利於活性分子的富集。
附圖說明
圖1為本發明流程圖;
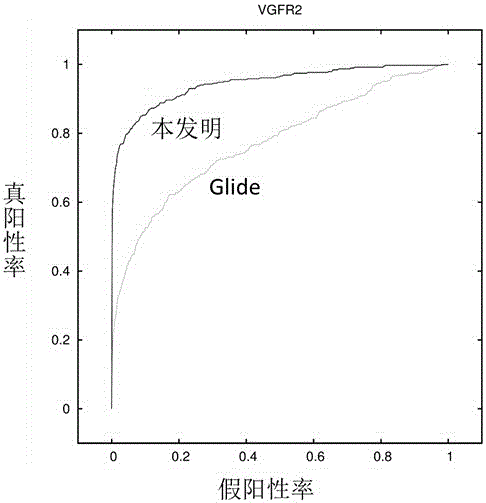
圖2為本發明針對靶標VGFR2的Glide與本發明結果的ROC評估圖;其中深色實線為本發明對應的受試者工作特徵曲線;淺色實線為由Glide分子對接得到的受試者工作特徵曲線;
圖3為本發明針對靶標VGFR2的Glide與PLEIC-SVM結果的EF評估圖;其中虛線為理想的富集曲線,淺色實線為Glide的結果得到的富集曲線,深色實線為利用本發明得到的富集曲線。
具體實施方式
本發明的具體步驟:
(1)從CHEMBL、BindingDB、DUD-E等資料庫中提取特定靶標的活性數據。要求活性小分子的IC50、Kd、Ki等活性數據小於10μM,非活性小分子從ZINC資料庫中提取。
(2)對活性分子和非活性分子進行相似性分析,以保證活性與非活性數據的多樣性。要求活性小分子間的ECFP4相似性小於0.8。非活性小分子是以活性小分子為模板挑選的,要求兩者物理上相似但化學性質上不相似。以每個活性分子為模板,尋找與每個活性小分子的分子質量之差不大於20,總的重原子數之差小於2,可旋轉鍵的數目之差不大於1,氫鍵供體的數目之差不大於1,氫鍵受體的數目之差不大於2,脂水分配係數之差不大於1的小分子。非活性小分子間的相似性小於0.4,非活性小分子與活性小分子的ECFP4相似性小於0.6。最後得到的活性小分子的數目大於100,非活性小分子的數目是活性小分子數目的50倍。
(3)分子對接。利用薛丁格分子對接軟體進行分子對接,每個小分子只保留打分最好的構象。
(4)計算找出結合口袋附近的胺基酸殘基。選定靶標PDB自身配體周圍範圍內的殘基作為候選殘基,以保證可以包圍絕大部分的小分子。
(5)計算每個小分子與結合口袋中的胺基酸殘基的相互作用能,形成相互作用能矩陣。計算對接後每個小分子與胺基酸殘基的範德華、氫鍵和疏水相互作用。
(6)統計相互作用能矩陣中每個元素出現的概率,去除出現次數較少的元素,形成相互作用指紋。如果相互作用矩陣中某一元素出現的頻率小於0.1那麼去除該元素。
(7)生成支持向量機輸入文件,利用網格搜索和交叉驗證尋找最優參數。
(8)利用(7)中得到的最優參數,交叉驗證評估模型。在此利用富集因子和受試者工作特徵曲線對模型進行評估。
(9)利用(7)中得到的最優參數,訓練全部樣本,得到篩選模型。
(10)利用篩選模型進行虛擬篩選。
實施例
結合附圖以建立VGFR2靶標的篩選模型為例對本發明進行詳細說明。
參閱圖1,首先要做的是,更改SVM軟體libsvm中的評價指標。從libsvm官方網站下載eval.cpp,eval.h,重新編譯,將網格搜索和交叉驗證的評估標準改為AUC。
(1)從DUD-E庫中收集VGFR2的活性數據,其中包含409個活性小分子,24950個非活性小分子。PDB文件為2P2I。
(2)計算2P2I中自身配體的中心坐標,(38,35,12)。
(3)利用薛丁格分子對接軟體Glide進行分子對接。
(4)對接後的每個分子只取GlideScore分數最低的構象。利用glide_ensemble_merge和glide_sort工具實現這一目的。
(5)將分子對接得到的小分子分開,放到文件夾mols裡面。
(6)計算找出參考分子以內的胺基酸殘基。一共包含60個殘基。
(7)對結合口袋的胺基酸殘基原子進行歸類,氫鍵供體、氫鍵受體、疏水原子。計算mols文件夾中每個小分子的原子特徵,並計算每個小分子與胺基酸殘基的相互作用能,範德華相互作用能、氫鍵相互作用能、疏水相互作用能。所有的相互作用能信息以每個分子一行的形式追到文件fingers.dat中。
(8)統計不同相互作用能元素出現的頻率,刪除出現頻率較小的元素。生成PLIEIC相互作用能指紋信息,存放到svm.dat中。最終保留了88維信息。
(9)生成支持向量機輸入文件,利用libsvm軟體工具包中的grid.py工具進行5折的交叉驗證和網格搜索。在本實施例中取C等於8,gamma等於1。
(10)利用(9)中得到的最優參數,利用受試者工作特徵曲線通過交叉驗證評估模型。結果如圖2所示,其中黑色的實線表示本發明對應的受試者工作特徵曲線,灰色的線表示由Glide分子對接得到的受試者工作特徵曲線。由圖可以看出,本發明對應的結果得到的受試者工作曲線下的面積(AUC)大於由Glide分子對接得到的AUC值,由此可以得出本發明可以降低假陽性率。
(11)利用(9)中得到的最優參數,利用富集因子通過交叉驗證評估模型。首先,根據富集因子的定義,得出理想的EF曲線;然後,對Glide得到的結果進行排序,得到Glide的EF曲線;最後對SVM得出的「可能性」(probability)進行排序,得到本發明對應的EF曲線。結果如圖3所示。其中黑色的虛線表示理想的富集曲線,灰色的實線代表Glide的結果得到的富集曲線,黑色的實線代表利用本發明得到的富集曲線。從圖中可以看出,利用PLEIC-SVM得到的曲線更接近於理想的富集曲線,即更有利於活性分子的富集。如果以佔數據集百分之一的比例為截斷,理想結果的富集因子為52,Glide的結果對應的富集因子為22,本發明的結果對應的富集因子為47,結果表明本發明可以使活性分子排在比較靠前的位置,提高活性分子的富集。
